一种细胞特异性基因转录水平的分析方法
作者:吴丽娟* 黎燕 沈倍奋
单位:丽娟 第三军医大学附属大坪医院野战外科研究所检验科 重庆,400042;黎燕 沈倍奋 军事医学科学院基础医学研究所分子免疫室 北京,100850;吴丽娟*,女,33岁,主治医师
关键词:细胞核;基因表达;mRNA
第三军医大学学报990614 提 要 目的:报道一种更为灵敏的细胞特异性基因转录水平分析方法。方法:用灵敏度、重复性和线性试验对该法进行评价。结果:本法可以对5×105的细胞进行检测;5×106的细胞受试,杂交信号强度CV值为0.11;在5×105~1×107细胞范围内,杂交信号强度与细胞数的线性关系较好。结论:该法灵敏度高,重复性和线性满意,可用于细胞核内新生成mRNA的分析。
, http://www.100md.com
中图法分类号 394-33
An analytic method for cellular specific gene transcription
Wu Lijuan, Li Yan, Shen Beifen (Department of Laboratory Tests, Daping Hosptital, Third Military Medical University, Chongqing,400042)
Abstract Objective: To report a method to analyze the cellular specific gene transcription. Methods: The sensitivity, reproducibility and linearity of the method were estimated. Results: 1×105 cells were able to gain a distinguishable positive hybridization signal. When the number of the cells was increased to 5×105 the hybridization signal was (10.00±1.12)% with a CV value of 0.11. The hybridization signal will further increase when the number of cells reaches to the range from 5×105 to 1×107. Conclusion: This analytic method possesses high sensitivity and satisfactory reproducibility and lenearity and can be used to analyze cellular specific gene transcription.
, 百拇医药
Key words cell nucleus; gene expressing; mRNA
1978年Marzluff WF等[1]描述了一种针对细胞核内新生成的mRNA(Newly transeribed RNA)进行分析的方法,即Nuclear runoff transcription assay,简称NRTA。由于该法依赖蔗糖梯度超速离心分离细胞核,以及靶mRNA分离纯化步骤繁琐,并未引起人们的重视。90年代后,随着细胞核及RNA纯化技术的日新月异,该技术重新得以重视并发展。我室调研了当前国外的报道,重建了NRTA,并使其灵敏度有所提高,现报道如下。
1 材料与方法
1.1 基本原理
NRTA的原理见图1。细胞在时间T1处经某种处理,基因A的转录水平逐渐增强,而基因B的转录水平无明显变化。在时间T1和T2收集细胞,用RT-PCR或Northern印迹技术分析基因A和基因B的转录水平时,试验提取的是细胞的总RNA,经浓度测定后,吸取少量(定量)的总RNA作为模板进行RT-PCR或使其吸附在硝酸纤维素膜上形成潜斑。在一定量的总RNA中,A基因转录产物增多,则B基因转录产物相对减少,所以,RT-PCR、Northern印迹反映的是待检基因累积性转录产物在当时总基因产物中的丰度,势必得到结论:从T1~T2基因A的转录水平是升高的,基因B的转录水平是降低的,可见基因B的描述不客观。NRTA技术在冰浴情况下立即分离的是某一“瞬时”的细胞核(要求该核与细胞质成份彻底分离而又完整无损,原有的生物学活性不受改变),在外界只提供转录所需的NTPs的情况下,依赖原有的转录酶促系统,将核分离瞬时暂停的转录继续进行下去并至完成。收集全部的新生mRNA与已知的基因A和基因B的DNA固相探针进行反相杂交,这里要求已知的DNA探针用量足够多,能够与液相中的全部新生成mRNA杂交。试验再对该新生成的同位素标记mRNA进行检测。由于新分离的细胞核内原有已完全转录生成的mRNA量少,且原始累积性mRNA对后续实验生成的同位素标记mRNA的放射自显影检测无影响,因此,NRTA能客观反映当时核内某特定基因的转录水平,代表着当时核内催化该特定基因转录的RNA聚合酶的活性,从而真实反映了当时细胞所处的功能状态。
, http://www.100md.com
图1 NRTA原理示意图
Fig 1 The diagrams of NRTA principle
1.2 cDNA固相探针的制备
应用PCR制备IL-6特异性cDNA片段(500 bp),经纯化后在90 μlcDNA水溶液中(含cDNA 600 ng)加入1mmol/L NaOH 10 μl,室温变性30 min,加入6×SSC 1 ml,冰浴中和,用6×SSC抽滤3次,每孔加入变性cDNA 100 ng,抽干,再用1ml 6×SSC清洗2次。将0.1% DEPC处理后的硝酸纤维素膜转入6×SSC浸泡2h,准备好狭缝装置,先用铅笔编号。空气过夜干燥,80°C烤2h,室温保存备用,临用前按需剪成小条。
1.3 细胞核的提取
收获SKO-007细胞,立即用冰预冷的1×PBS洗涤2次,在5×106细胞沉淀中加入预冷的NP-40裂解液[10 mmol/L Tris-HCl(pH7.9),100 mmol/L NaCl,1 mmol/L EDTA, 0.5% NP-40]1.0 ml,温和混匀,冰浴10 min,4°C以500×g离心5 min,吸弃上清液。用冰预冷1×PBS同法洗涤一次,向细胞核沉淀中加入100 μl冰预冷甘油缓冲液[50 mmol/L Tris-HCL (pH8.3), 5 mmol/L MgCl2, 100 mmol/L EDTA, 40%甘油],用吸管轻轻吹吸混匀,立即置入-70°C备用。
, 百拇医药
1.4 新生转录反应
室温解冻细胞核,立即加入100 μl2倍反应缓冲液[10 mmol/L Tris-HCl (pH8.0), 5 mmol/L MgCl2,300 mmol/L KCl, 1 mmol/L ATP, 1 mmol/L CTP, 1 mmol/L GTP, 5 mmol/L DTT]和100 μCi[α-32P]UTP(购于北京亚辉生物医学工程公司,批号199704027),轻轻吹吸混匀,立即置25°C水浴,振摇反应60 min。
1.5 新生32P标记mRNA的提取
操作同文献[3]。将得到的RNA溶于100 μlTES溶液[10 mmol/L Tris(base)(pH7.4),10 mmol/L EDTA, 0.2% SDS]中。
, 百拇医药
1.6 RNA与cDNA的杂交反应
向杂交反应管补加100 μlTES/NaCl溶液[10 mmol/L Tris(base)(pH7.4),10 mmol/L EDTA,0.2% SDS,0.6 mmol/L NaCl],混匀后放入cDNA硝酸纤维素膜条,用胶布将管盖扎紧,立即没入65°C水浴摇床中杂交36 h。取出膜条放入一个烧杯中,用25 ml 2×SSC 65°C洗膜3~4次,每次30 min。将硝酸纤维素膜投入盛1.5 ml 2×SSC/8 μl5mg/ml RNaseA的试管内,室温静置40 min。用25 ml 2×SSC室温洗膜3~4次,每次15 min。取出膜条,用透明胶将其固定在滤纸上。压片-70°C暴光3~4d,放射自显影后,用激光扫描仪测相对杂交信号强度。
2 结果
2.1 灵敏度
, http://www.100md.com
用1×105细胞实验即可得到肉眼可分辩的阳性杂交性号,5×105以上的细胞阳性杂交信号明确,见图2。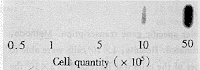
图2 灵敏度试验结果
Fig 2 The results of sensitivity test by the same group cultured SKO-007 cells
用5×104~5×106同批稳态培养的SKO-007细胞进行试验,结果5×105细胞阳性杂交信号明确。
2.2 重复试验
将同批收获的SKO-007细胞等分成10份,每份5×106细胞,结果杂交信号强度为(10.00±1.12)%,CV值0.11,见图3。
, http://www.100md.com
图3 重复性试验结果
Fig 3 The results of repeativity test by 10 portions of the same group cultured SKO-007 cells at 5×106
用10份同批培养的5×106 SKO 007细胞试验,结果杂交信号强度相差不大。
2.3 线性范围
细胞数在5×105~1×107范围内随着细胞用量的增加杂交信号加强,线性良好,见图4。
图4 线性试验结果
, http://www.100md.com
Fig 4 Relationship between cell quantity and relative signal intensity of hybridization
3 讨论
NRTA在近年国外一些有关的文献报道中已有不少应用的实例,其实验方案大体一致,只是在细胞核提取和同位素标记mRNA的提取方法上有一些差异。值得注意的是:细胞核的提取是NRTA技术的关键步骤,直接影响新生转录中32P标记RNA的掺入率。国外文献报道的核提取方法较多,如蔗糖梯度超速离心法[1]、Donuce匀浆法[4]、低渗缓冲液裂解法[5],NP-40裂解法[2]等。其中NP-40裂解法操作简单、快速 ,为当前大多数学者采用,我们将配方中Tirs-HCL的pH调至7.9,将NaCl浓度调至100 mmol/L,用0.5%NP-40经冰浴5 min的反应,细胞裂解彻底,所收获的核胞质成份去除彻底,体积无明显改变,核膜完整,核仁清晰,实验最终得到的杂交信号最强。
, http://www.100md.com
新生成mRNA的提取是影响方法灵敏度的重要环节。大多数文献采用AGPC法[6],也可见盐酸胍/氯化铯法[8],以及以tRNA为载体的蛋白酶K/TCA沉淀法[1]。我们将细菌RNA制备用的SDS/Phenol法改进后用于新生mRNA提取,实验结果满意[3]。
另外,国外文献报道的新生转录反应温度为42°C,反应时间30~40 min,(1~5)×107的细胞[α-32P]UTP用量在50~250μCi。我们的研究提示:最佳反应温度应为25°C,反应时间60 min, 5×106的细胞[α-32P]UTP用量为100μCi。同时,我们用PCR克隆和经纯化的cDNA片段代替全质粒探针实验,经上述处理后,使方法的灵敏度提高了十倍,受试细胞数(1~5)×107下调至(1~5)×106。
, http://www.100md.com
NRTA为细胞特异性基因转录分析方法,其特点是提取一份RNA可以同时对几种,甚致十余种特异性基因的转录水平同时进行分析,大大减少了操作,而RT-PCR、Northern印迹等方法一次测定只能针对一种特定基因。另外,NRTA是对核内新生成的mRNA进行分析,排除了存于细胞质中的大量未即时翻译mRNA的影响。NRTA一次试验的结果排列在同一纸条上,排除了RT-PCR、Northern印迹等因多管测定而可能存在的多次加样等偶然因素的影响,结果的可比性强。
总之,NRTA特别适合于分析一种信号传导引起的细胞一组特异基因在转录水平上的变化,是一种很有前途的基因转录分析方法。
(本课题承蒙沈倍奋院士、黎燕老师的带教和指导,以及倪长源、胡美茹、王建安等老师的多方帮助,在此一并表示感谢!)
参考文献
, 百拇医药 1 Marzluff W F. Transcription of RNA in isolated nuclei. Methods Cell Biol,1978,19:317
2 Robert J S, Trek S K, Philip A M. Vascular platet endothelial cell adhesion molecule-1 (PECAM-1) expression is decreased by TNF-αand TNF-γ.J Immunol,1996,156(3):1221
3 吴丽娟,黎 燕,沈倍奋.改进SDS-Phenol法快速提取细胞总RNA.第三军医大学学报,1999,21(1):57
4 Look D L, Pelletier M R, Holtzman M T. Selective interaction of a subset of Interferon-γ response element-binding proteins with the intercelluar adhesion molecule-1 (ICAM-1) gene promoter controls the pattern of expression on epithelial cell. J Biol Chem,1994,269(12):8952
, 百拇医药
5 Satterthwaite A B, Borson R, Tenen D G. Regulation of the gene for CD34, a human hematopoietic stem cell antigen, in KG-1 cells. Blood,1990,75(12):2299
6 Naora H, Young I G. Comparison of the mechnisms regulting IL-5, IL-4,and three other lymphokine genes in the Th2 clone D10-G4-1. Exp Hematol,1995,23(7):567
7 Takaishi T, Matsui T, Tsukamotm T, et al. TGF-β-induced macrophage colony-stimulating factor gene expression in various mesenchymal cell lines. Am J Physiol,1994,267(1 pt1):C25
8 Colotta F, Fabio R, Muzio M, et al. Interleukin-13 induces expression and release of Interleukin-1 decoy receptor in human polymorphonuclear cells. J Biol Chem,1994,269(17):12403
收稿:1998-10-16;修回:1999-01-26, http://www.100md.com(吴丽娟* 黎燕 沈倍奋)
单位:丽娟 第三军医大学附属大坪医院野战外科研究所检验科 重庆,400042;黎燕 沈倍奋 军事医学科学院基础医学研究所分子免疫室 北京,100850;吴丽娟*,女,33岁,主治医师
关键词:细胞核;基因表达;mRNA
第三军医大学学报990614 提 要 目的:报道一种更为灵敏的细胞特异性基因转录水平分析方法。方法:用灵敏度、重复性和线性试验对该法进行评价。结果:本法可以对5×105的细胞进行检测;5×106的细胞受试,杂交信号强度CV值为0.11;在5×105~1×107细胞范围内,杂交信号强度与细胞数的线性关系较好。结论:该法灵敏度高,重复性和线性满意,可用于细胞核内新生成mRNA的分析。
, http://www.100md.com
中图法分类号 394-33
An analytic method for cellular specific gene transcription
Wu Lijuan, Li Yan, Shen Beifen (Department of Laboratory Tests, Daping Hosptital, Third Military Medical University, Chongqing,400042)
Abstract Objective: To report a method to analyze the cellular specific gene transcription. Methods: The sensitivity, reproducibility and linearity of the method were estimated. Results: 1×105 cells were able to gain a distinguishable positive hybridization signal. When the number of the cells was increased to 5×105 the hybridization signal was (10.00±1.12)% with a CV value of 0.11. The hybridization signal will further increase when the number of cells reaches to the range from 5×105 to 1×107. Conclusion: This analytic method possesses high sensitivity and satisfactory reproducibility and lenearity and can be used to analyze cellular specific gene transcription.
, 百拇医药
Key words cell nucleus; gene expressing; mRNA
1978年Marzluff WF等[1]描述了一种针对细胞核内新生成的mRNA(Newly transeribed RNA)进行分析的方法,即Nuclear runoff transcription assay,简称NRTA。由于该法依赖蔗糖梯度超速离心分离细胞核,以及靶mRNA分离纯化步骤繁琐,并未引起人们的重视。90年代后,随着细胞核及RNA纯化技术的日新月异,该技术重新得以重视并发展。我室调研了当前国外的报道,重建了NRTA,并使其灵敏度有所提高,现报道如下。
1 材料与方法
1.1 基本原理
NRTA的原理见图1。细胞在时间T1处经某种处理,基因A的转录水平逐渐增强,而基因B的转录水平无明显变化。在时间T1和T2收集细胞,用RT-PCR或Northern印迹技术分析基因A和基因B的转录水平时,试验提取的是细胞的总RNA,经浓度测定后,吸取少量(定量)的总RNA作为模板进行RT-PCR或使其吸附在硝酸纤维素膜上形成潜斑。在一定量的总RNA中,A基因转录产物增多,则B基因转录产物相对减少,所以,RT-PCR、Northern印迹反映的是待检基因累积性转录产物在当时总基因产物中的丰度,势必得到结论:从T1~T2基因A的转录水平是升高的,基因B的转录水平是降低的,可见基因B的描述不客观。NRTA技术在冰浴情况下立即分离的是某一“瞬时”的细胞核(要求该核与细胞质成份彻底分离而又完整无损,原有的生物学活性不受改变),在外界只提供转录所需的NTPs的情况下,依赖原有的转录酶促系统,将核分离瞬时暂停的转录继续进行下去并至完成。收集全部的新生mRNA与已知的基因A和基因B的DNA固相探针进行反相杂交,这里要求已知的DNA探针用量足够多,能够与液相中的全部新生成mRNA杂交。试验再对该新生成的同位素标记mRNA进行检测。由于新分离的细胞核内原有已完全转录生成的mRNA量少,且原始累积性mRNA对后续实验生成的同位素标记mRNA的放射自显影检测无影响,因此,NRTA能客观反映当时核内某特定基因的转录水平,代表着当时核内催化该特定基因转录的RNA聚合酶的活性,从而真实反映了当时细胞所处的功能状态。

, http://www.100md.com
图1 NRTA原理示意图
Fig 1 The diagrams of NRTA principle
1.2 cDNA固相探针的制备
应用PCR制备IL-6特异性cDNA片段(500 bp),经纯化后在90 μlcDNA水溶液中(含cDNA 600 ng)加入1mmol/L NaOH 10 μl,室温变性30 min,加入6×SSC 1 ml,冰浴中和,用6×SSC抽滤3次,每孔加入变性cDNA 100 ng,抽干,再用1ml 6×SSC清洗2次。将0.1% DEPC处理后的硝酸纤维素膜转入6×SSC浸泡2h,准备好狭缝装置,先用铅笔编号。空气过夜干燥,80°C烤2h,室温保存备用,临用前按需剪成小条。
1.3 细胞核的提取
收获SKO-007细胞,立即用冰预冷的1×PBS洗涤2次,在5×106细胞沉淀中加入预冷的NP-40裂解液[10 mmol/L Tris-HCl(pH7.9),100 mmol/L NaCl,1 mmol/L EDTA, 0.5% NP-40]1.0 ml,温和混匀,冰浴10 min,4°C以500×g离心5 min,吸弃上清液。用冰预冷1×PBS同法洗涤一次,向细胞核沉淀中加入100 μl冰预冷甘油缓冲液[50 mmol/L Tris-HCL (pH8.3), 5 mmol/L MgCl2, 100 mmol/L EDTA, 40%甘油],用吸管轻轻吹吸混匀,立即置入-70°C备用。
, 百拇医药
1.4 新生转录反应
室温解冻细胞核,立即加入100 μl2倍反应缓冲液[10 mmol/L Tris-HCl (pH8.0), 5 mmol/L MgCl2,300 mmol/L KCl, 1 mmol/L ATP, 1 mmol/L CTP, 1 mmol/L GTP, 5 mmol/L DTT]和100 μCi[α-32P]UTP(购于北京亚辉生物医学工程公司,批号199704027),轻轻吹吸混匀,立即置25°C水浴,振摇反应60 min。
1.5 新生32P标记mRNA的提取
操作同文献[3]。将得到的RNA溶于100 μlTES溶液[10 mmol/L Tris(base)(pH7.4),10 mmol/L EDTA, 0.2% SDS]中。
, 百拇医药
1.6 RNA与cDNA的杂交反应
向杂交反应管补加100 μlTES/NaCl溶液[10 mmol/L Tris(base)(pH7.4),10 mmol/L EDTA,0.2% SDS,0.6 mmol/L NaCl],混匀后放入cDNA硝酸纤维素膜条,用胶布将管盖扎紧,立即没入65°C水浴摇床中杂交36 h。取出膜条放入一个烧杯中,用25 ml 2×SSC 65°C洗膜3~4次,每次30 min。将硝酸纤维素膜投入盛1.5 ml 2×SSC/8 μl5mg/ml RNaseA的试管内,室温静置40 min。用25 ml 2×SSC室温洗膜3~4次,每次15 min。取出膜条,用透明胶将其固定在滤纸上。压片-70°C暴光3~4d,放射自显影后,用激光扫描仪测相对杂交信号强度。
2 结果
2.1 灵敏度
, http://www.100md.com
用1×105细胞实验即可得到肉眼可分辩的阳性杂交性号,5×105以上的细胞阳性杂交信号明确,见图2。
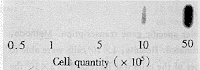
图2 灵敏度试验结果
Fig 2 The results of sensitivity test by the same group cultured SKO-007 cells
用5×104~5×106同批稳态培养的SKO-007细胞进行试验,结果5×105细胞阳性杂交信号明确。
2.2 重复试验
将同批收获的SKO-007细胞等分成10份,每份5×106细胞,结果杂交信号强度为(10.00±1.12)%,CV值0.11,见图3。

, http://www.100md.com
图3 重复性试验结果
Fig 3 The results of repeativity test by 10 portions of the same group cultured SKO-007 cells at 5×106
用10份同批培养的5×106 SKO 007细胞试验,结果杂交信号强度相差不大。
2.3 线性范围
细胞数在5×105~1×107范围内随着细胞用量的增加杂交信号加强,线性良好,见图4。

图4 线性试验结果
, http://www.100md.com
Fig 4 Relationship between cell quantity and relative signal intensity of hybridization
3 讨论
NRTA在近年国外一些有关的文献报道中已有不少应用的实例,其实验方案大体一致,只是在细胞核提取和同位素标记mRNA的提取方法上有一些差异。值得注意的是:细胞核的提取是NRTA技术的关键步骤,直接影响新生转录中32P标记RNA的掺入率。国外文献报道的核提取方法较多,如蔗糖梯度超速离心法[1]、Donuce匀浆法[4]、低渗缓冲液裂解法[5],NP-40裂解法[2]等。其中NP-40裂解法操作简单、快速 ,为当前大多数学者采用,我们将配方中Tirs-HCL的pH调至7.9,将NaCl浓度调至100 mmol/L,用0.5%NP-40经冰浴5 min的反应,细胞裂解彻底,所收获的核胞质成份去除彻底,体积无明显改变,核膜完整,核仁清晰,实验最终得到的杂交信号最强。
, http://www.100md.com
新生成mRNA的提取是影响方法灵敏度的重要环节。大多数文献采用AGPC法[6],也可见盐酸胍/氯化铯法[8],以及以tRNA为载体的蛋白酶K/TCA沉淀法[1]。我们将细菌RNA制备用的SDS/Phenol法改进后用于新生mRNA提取,实验结果满意[3]。
另外,国外文献报道的新生转录反应温度为42°C,反应时间30~40 min,(1~5)×107的细胞[α-32P]UTP用量在50~250μCi。我们的研究提示:最佳反应温度应为25°C,反应时间60 min, 5×106的细胞[α-32P]UTP用量为100μCi。同时,我们用PCR克隆和经纯化的cDNA片段代替全质粒探针实验,经上述处理后,使方法的灵敏度提高了十倍,受试细胞数(1~5)×107下调至(1~5)×106。
, http://www.100md.com
NRTA为细胞特异性基因转录分析方法,其特点是提取一份RNA可以同时对几种,甚致十余种特异性基因的转录水平同时进行分析,大大减少了操作,而RT-PCR、Northern印迹等方法一次测定只能针对一种特定基因。另外,NRTA是对核内新生成的mRNA进行分析,排除了存于细胞质中的大量未即时翻译mRNA的影响。NRTA一次试验的结果排列在同一纸条上,排除了RT-PCR、Northern印迹等因多管测定而可能存在的多次加样等偶然因素的影响,结果的可比性强。
总之,NRTA特别适合于分析一种信号传导引起的细胞一组特异基因在转录水平上的变化,是一种很有前途的基因转录分析方法。
(本课题承蒙沈倍奋院士、黎燕老师的带教和指导,以及倪长源、胡美茹、王建安等老师的多方帮助,在此一并表示感谢!)
参考文献
, 百拇医药 1 Marzluff W F. Transcription of RNA in isolated nuclei. Methods Cell Biol,1978,19:317
2 Robert J S, Trek S K, Philip A M. Vascular platet endothelial cell adhesion molecule-1 (PECAM-1) expression is decreased by TNF-αand TNF-γ.J Immunol,1996,156(3):1221
3 吴丽娟,黎 燕,沈倍奋.改进SDS-Phenol法快速提取细胞总RNA.第三军医大学学报,1999,21(1):57
4 Look D L, Pelletier M R, Holtzman M T. Selective interaction of a subset of Interferon-γ response element-binding proteins with the intercelluar adhesion molecule-1 (ICAM-1) gene promoter controls the pattern of expression on epithelial cell. J Biol Chem,1994,269(12):8952
, 百拇医药
5 Satterthwaite A B, Borson R, Tenen D G. Regulation of the gene for CD34, a human hematopoietic stem cell antigen, in KG-1 cells. Blood,1990,75(12):2299
6 Naora H, Young I G. Comparison of the mechnisms regulting IL-5, IL-4,and three other lymphokine genes in the Th2 clone D10-G4-1. Exp Hematol,1995,23(7):567
7 Takaishi T, Matsui T, Tsukamotm T, et al. TGF-β-induced macrophage colony-stimulating factor gene expression in various mesenchymal cell lines. Am J Physiol,1994,267(1 pt1):C25
8 Colotta F, Fabio R, Muzio M, et al. Interleukin-13 induces expression and release of Interleukin-1 decoy receptor in human polymorphonuclear cells. J Biol Chem,1994,269(17):12403
收稿:1998-10-16;修回:1999-01-26, http://www.100md.com(吴丽娟* 黎燕 沈倍奋)