用mRNA差异显示法分离大鼠吗啡依赖相关基因
作者:崔振中 梁德勇 金蕾 罗非 王晓民 韩济生
单位:北京医科大学神经科学研究所,北京 100083
关键词:吗啡依赖;基因扩增;RNA;信使;代谢;DNA;环状;代谢
990307 摘 要 目的:分离并克隆大鼠吗啡依赖相关基因。方法:SD大鼠用递增剂量的吗啡作皮下注射,形成吗啡依赖的动物模型。分离鼠脑的中脑导水管周围灰质、伏核、纹状体等核团,提取总RNA,用mRNA差异显示(differential display PCR, DDPCR)的方法,筛选吗啡依赖大鼠特异表达产物的cDNA片段。用3组锚定引物(T12MN,M=A,G,T或C,N=A,C 或G)和6组随机引物(AP1~AP6)作不同组合进行PCR扩增。结果:获得了80余个差异表达产物的cDNA片段,经斑点杂交初步筛选,克隆了10个阳性片段,选取4个克隆进行序列分析,获得了4个cDNA片段。结论:通过BLAST数据库序列比较,现已确认获得了4个吗啡依赖表达的新基因片段。
, http://www.100md.com
中国图书资料分类法分类号 Q343.12-332
A preliminary study on the isolation of specially expressed genes
of morphine dependent rat by differential display PCR
CUI Zhen-Zhong, LIANG De-Yong, JIN Lei, LUO Fei, WANG Xiao-Min, HAN Ji-Sheng
(Neuroscience Research Institute, Beijing Medical University, Beijing 100083)
MeSH Gene amplification Morphine dependence RNA, messenger/metab DNA, circular/metab
, 百拇医药
ABSTRACT Objective: To isolate and clone morphine dependent genes of SD rat. Methods: SD rats were used for the morphine dependent model. They were injected with increasing dose of morphine for 10 days. At single inject, the dose of morphine was from 5 mg/kg to 120 mg/kg. The rats were then scarified and the brain region was disassociated. The nucleus such as periaqueductal gray (PAG), nucleus accumbens, striatum were selected for total RNA extraction. Differential display-PCR (DD-PCR) of mRNA was used for isolating the specific expressed genes. Three sets of anchor primers (T12MN, M=A,G,T or C; N=A,C or G) and 6 sets of arbitrary primers(AP1~AP6) were used for DDPCR amplification. By combination of anchor primers and arbitrary primers, series of PCR reaction were preformed. Results: More than 80 cDNA fragments, which might represent the differentially expressed genes, were obtained by DDPCR method. Dot blot was used for further selection of positive cDNA, and more than 10 cDNA fragments with strong positive signals were cloned. Four clones were sequenced. Four cDNA fragments were obtained. Conclusion: Based on the sequence homologous comparison with NIH BLAST data, four specially expressed new gene fragments have been isolated and cloned by using mRNA differential display-PCR (DDPCR) method.
, 百拇医药
(J Beijing Med Unvi, 1999,31:216-219)
mRNA差异显示技术(differential display PCR,DDPCR)是1992年由Liang等[1]建立,在筛选差异表达基因及新基因分离方面发挥了重要的作用[2,3]。在神经科学领域,DDPCR方法也有广泛的应用[4]。药物依赖是一个重要的研究课题,近年的基础研究业已发现一些重要的基因,如fos、jun、多巴胺受体及creb等基因在药物依赖的过程中起作用[5],但对药物成瘾的机制还远未明了。应用DDPCR的方法,直接从基因表达水平寻找并获得差异表达基因,进行功能分析,探讨其在药物依赖过程中的作用,将是一个有意义的研究方向。
1 材料与方法
1.1 材料
, 百拇医药
SD大鼠,雄性,250g,2月龄,购自北京医科大学实验动物中心;RNA提取试剂,购自GIBCO公司;DDPCR引物由Sybesyn公司合成,锚定引物(3组):,T12MN(M=A,T,G,C;N=A,C,G),随机引物(6组):AP1~AP6;TaqDNA聚合酶,逆转录酶(MMLV-RT),E.coliDNA聚合酶,E.coliDNA连接酶,T4DNA连接酶,购自BoehringerMannheim;RNasin,购自Promega;mRNA纯化试剂盒,购自Phamacia;cDNA合成试剂盒,cDNA消减杂交试剂盒,cDNA克隆试剂盒,购自Clontech;α-p32-dATP,购自北京亚辉生物医学公司。
1.2 实验大鼠的处理
大鼠分为实验组和对照组。实验组大鼠皮下注射递增剂量的吗啡:第1天5mg.kg-1,第2天10mg.kg-1,第3、4天20mg.kg-1,第5、6天40mg.kg-1,第7、8天80mg.kg-1,第9、10天120mg.kg-1;每天2次,连续注射7~10d,直至出现依赖症状。对照组大鼠注射同等剂量的生理盐水。最后一次注射后4h将大鼠断头,开颅,迅速切取大鼠的中脑导水管周围灰质(PAG)、伏核、纹状体,用于总RNA提取。
, 百拇医药
1.3 总RNA的提取
参考硫氰酸胍一步法稍加改进,取1mlRNA提取试剂,加入玻璃匀浆器中,将大鼠的脑组织置于匀浆器中,制备匀浆,转入1.5ml小离心管中,加100μl氯仿,于4℃,15000r.min-1,离心15min,取上清水相,转入新的离心管中,加等体积异丙醇沉淀RNA。RNA重溶于50μl水中,加10单位的RNasin,取2μlRNA样品进行琼脂糖凝胶电泳,检测RNA的完整性,取1μl进行紫外分光光度法检测RNA的浓度和纯度。其余样品-80℃保存。
1.4 逆转录反应
取3μg总RNA样品,加5单位无RNA酶活性的DNaseI消化样品中残存的DNA,在10μl体积下37℃保温20min,65℃保温5min,灭活DNaseI活性。加5单位RNasin,4μl5×1stBuffer,1μl10mmol.L-1dNTPs,1μloligodT锚定引物(T12G,或T12A,或T12C),5单位MMLV-RT逆转录酶,加水至20μl,37℃保温1h,65℃保温5min,灭活逆转录酶活性,样品可于-20℃存放。
, 百拇医药
1.5 PCR扩增
用3套锚定引物与6种(AP1~AP6)随机引物作不同组合,进行18个PCR反应,PCR反应所用的锚定引物应与逆转录的锚定引物相同。依次加入下列样品:逆转录产物2μl,10×PCRBuffer2.5μl,25mmol.L-1MgCl21.5μl,随机引物1μl,锚定引物1μl,10mmol.L-1dNTPs1 μl,TaqDNA聚合酶1μl,加水至25μl,滴加30μl石蜡油,按下列条件进行PCR扩增:94℃2min;94℃40s,40℃50s,72℃1.5min,40个循环;72℃7min。PCR结束后,先用琼脂糖凝胶电泳检测PCR扩增的结果,若结果满意,用聚丙烯酰胺凝胶电泳分离差异条带。
1.6 聚丙烯酰胺凝胶电泳
, 百拇医药
配制80g.L-1含8mol.L-1尿素的聚丙烯酰胺变性凝胶,凝胶20cm×40cm,PCR产物加入适量的6×loadingbuffer,每一泳道加入约20μl样品,于800~1000V电压下电泳3~4h,上样时对照组和实验组加载于相邻泳道,以便于比较条带差异。
1.7 银染法显示差异条带
电泳结束后按下述条件进行银染。凝胶在100g.L-1醋酸中浸泡20min,水洗10min,于染色液(AgNO33g,12.2mol.L-1甲醛0.7ml,加水1L)中染色30min,水洗10s,于显色液(Na2CO330g,20g.L-1硫代硫酸钠200μl,12.2mol.L-1甲醛1.5ml,加水1L)中显色5~10min,显色后于100g.L-1醋酸中浸泡10min,保存凝胶、拍照、比较结果。
, 百拇医药
1.8 差异条带的第二次PCR扩增及克隆
切取差异条带,加入20μl水,70℃水浴20min,取1μl作模板用相同的引物在相同的条件下进行PCR扩增,低熔点琼脂糖凝胶电泳纯化回收目标条带。
1.9 差异序列的斑点杂交
将第二次PCR扩增片段加载于尼龙膜上与cDNA探针进行杂交,杂交条件参考分子克隆实验手册。探针的制备:取与DDPCR相同的RNA样品,经逆转录反应合成双链cDNA,纯化后用随机引物标记试剂盒标记探针,放射性同位素为α-p32-dCTP。选取阳性扩增片段克隆于ClontechTA载体中并进行序列分析。
1.10 序列分析及序列比较
将序列分析所获得的数据与GeneBank数据库资料进行比较。NCBI同源性比较的网址:WWW.ncbi.nlm.nih.gov。
, http://www.100md.com
2 结果
2.1 总RNA的提取
用RNA提取的标准程序提取了大鼠的PAG、伏核、纹状体等核团样本中的总RNA。大鼠伏核的总RNA电泳图谱见图1。所提取的RNA完整、未降解,符合实验要求,28S/18S的比值大于1.5,同时用紫外分光光度法对RNA的纯度及浓度进行了测定,其OD260/280的比值接近2.0。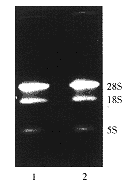
1, morphine treatment group; 2,control group. 5S, 18 S, 28 S RNA represent rRNA of total RNA, The higher ratio of 28S, the better quality of total RNA and its mRNA.
, 百拇医药
图1 伏核总RNA样品的电泳检测
Figure 1 Electrophoresis detection of total RNA sample
of N. Accumbens
2.2 吗啡依赖大鼠mRNA差异显示结果
经多轮次18组PCR扩增反应,获得了40余条差示条带,按1.7的方法对这些差示条带进行了第二次PCR扩增。图2为mRNA差异显示的图谱之一,图3是第二次PCR扩增的图谱。为了进一步鉴定这些差示条带,对这些差示条带进行了斑点杂交,初步克隆了10个杂交阳性片段。斑点杂交结果见图4。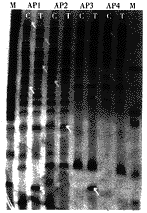
AP1~AP4, represents different arbitrary primers; C, control group; T, morphine treatment group; M,PCR marker. represents differentially expressed cDNA bands.
represents differentially expressed cDNA bands.
, 百拇医药
图2 银染法mRNA差异显示电泳图谱之一
Figure 2 A picture of the DDPCR amplification by silver staining
1~10 represent different cDNA fragment clones.
图3 差异表达产物的cDNA片段的第2轮PCR扩增
Figure 3 Second round PCR amplification
of differentially expressed cDNA fragments
, http://www.100md.com a, b, c, d represent cDNA fragments of pAC11, pAC12,pAC13, pAC14, respectively.
图4 斑点杂交结果
Figure 4 The result of dot blot
2.3 差示条带的序列分析及比较
对4个强阳性克隆进行了序列分析,并通过BLAST数据库进行了序列的同源性比较。比较结果显示这4个片段均为新基因片段,在数据库中查询未见同源序列。4个阳性片段的数据库查询部分结果是pAC11:Score=38, E Value=0.65; pAC12: Score=40, E Value=0.18; pAC13: Score=40。 E Value=0.24; pAC14: Score=40, E Value=0.54。Gene Bank登录情况为pAC11, AF145788; pAC12, AF145789; pAC13, AF145794; pAC14, AF145795, AF113006。图5列出了4个cDNA片段的序列。
, http://www.100md.com
图5 4个新cDNA片段的序列
Figure 5 The sequence of 4 novel cDNA fiagments
3 讨论
分离新基因的战略目前倍受重视,通过差异分析,获得在不同处理过程中的细胞或动物的特异表达的cDNA在目前是一个行之有效的方法,也比较符合我国国情。本研究利用DDPCR方法获得了4个差异表达产物的cDNA片段,通过Gene Bank数据库同源性比较,没有发现同源序列。下一步的主要工作是进行Northern杂交分析,预测全长的mRNA的长度;脑切片的原位杂交,分析其在脑内的表达情况;通过RACE扩增的方法,获得全长的cDNA,通过氨基酸序列推导预测其功能,并进行一系列的功能分析。
根据文献[6~8]报道及我们实验的体会,DDPCR技术虽然是行之有效的扩增新基因的方法,但也会产生大量的假阳性片段,这是一个难以克服的缺点。我们的解决办法是获得扩增片段后,先进行斑点杂交,标记cDNA探针进行同组杂交及交叉杂交(同组杂交:实验组探针同实验组片段杂交,对照组探针同对照组片段杂交;交叉杂交:实验组探针同对照组片段杂交;对照组片段同实验组片段杂交),来筛选较有意义的差异片段。对这些片段进行序列分析及比较,避免大规模测序造成的人力和财力的浪费。斑点杂交的方法有许多文献报道[9,10],我们的方法略有改进,这样更易于操作同时又能保证阳性结果。我们使用的探针是用作DDPCR相同的总RNA,通过逆转录反应合成cDNA的第一链和第二链,合成的cDNA基本上代表了样品中原有的mRNA序列,用随机引物方法标记探针,分别同获得的差异片段进行杂交,只挑选在同组杂交中阳性而交叉杂交阴性的片段作进一步分析。我们的实验结果证实这一方法是行之有效的,具有较强的可信度及可操作性,能有效地克服DDPCR的假阳性高、工作量大的缺点。我们同时还进行了用抑制性消减杂交分离吗啡依赖大鼠特异表达基因的研究工作,也取得了部分实验结果,将另文发表。
, 百拇医药
* 国家自然科学基金(39600192)资助项目。
参考文献
1 Liang P, Pardee AB. Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction. Science, 1992, 257:967-971
2 Hsu DKW, Donohue PJ, Alberts GF, et al. Fibroblast growth factor-1 induces phosphofructokinase fatty acid synthase and Ca2+-ATPase mRNA expression in NIH3T3 cells. Biochem Biophys Res Comm, 1993, 197:1483-1491
, 百拇医药
3 McClelland M, Mathieu-Daude F, Welsh J. Fingerprinting and differential display using arbitrary primed PCR. Trends Genet, 1995, 11:242-246
4 Babity JM, Newton RA, Guido ME, et al. The application of differential display to the brain: adaptations for the study of heterogeneous tissue. In: Liang P, Pardee AB, ed. Methods in Molecular Biology. Iotowa:Humana Press, 1997 (V85).285-295
5 Nestler EJ. Molecular mechanisms underlying opiate addication: implications of for medication development. Neuroscience, 1997, 9:84-93
, 百拇医药
6 Bertioli DJ, Schlichter UH, Adams MJ, et al. An analysis of differential display shows a strong bias towards high copy number mRNAs. Nucl Acids Res, 1995, 23:4520-4523
7 Debouck C. Differential display or differential dismay? Curr Opin Biotechnol, 1995, 6:597-599
8 Wadhwa R, Duncan E, Kaul SC, et al. An effective elimination of false positives isolated from differential display of mRNAs. Mol Biotechnol, 1996, 6:213-217
, 百拇医药
9 Liang P, Averboukh L, Pardee AB. Distribution and cloning of eukaryotic mRNAs by means of differential display: refinements and optimization. Nucl Acids Res, 1993, 21:3269-3275
10 Poirier GMC, Pyati J, Wan JS, et al. Screening differentially expressed cDNA clones obtained by differential display using amplified RNA. Nucl Acids Res, 1997, 25:913-914
(1999-03-09收稿), 百拇医药
单位:北京医科大学神经科学研究所,北京 100083
关键词:吗啡依赖;基因扩增;RNA;信使;代谢;DNA;环状;代谢
990307 摘 要 目的:分离并克隆大鼠吗啡依赖相关基因。方法:SD大鼠用递增剂量的吗啡作皮下注射,形成吗啡依赖的动物模型。分离鼠脑的中脑导水管周围灰质、伏核、纹状体等核团,提取总RNA,用mRNA差异显示(differential display PCR, DDPCR)的方法,筛选吗啡依赖大鼠特异表达产物的cDNA片段。用3组锚定引物(T12MN,M=A,G,T或C,N=A,C 或G)和6组随机引物(AP1~AP6)作不同组合进行PCR扩增。结果:获得了80余个差异表达产物的cDNA片段,经斑点杂交初步筛选,克隆了10个阳性片段,选取4个克隆进行序列分析,获得了4个cDNA片段。结论:通过BLAST数据库序列比较,现已确认获得了4个吗啡依赖表达的新基因片段。
, http://www.100md.com
中国图书资料分类法分类号 Q343.12-332
A preliminary study on the isolation of specially expressed genes
of morphine dependent rat by differential display PCR
CUI Zhen-Zhong, LIANG De-Yong, JIN Lei, LUO Fei, WANG Xiao-Min, HAN Ji-Sheng
(Neuroscience Research Institute, Beijing Medical University, Beijing 100083)
MeSH Gene amplification Morphine dependence RNA, messenger/metab DNA, circular/metab
, 百拇医药
ABSTRACT Objective: To isolate and clone morphine dependent genes of SD rat. Methods: SD rats were used for the morphine dependent model. They were injected with increasing dose of morphine for 10 days. At single inject, the dose of morphine was from 5 mg/kg to 120 mg/kg. The rats were then scarified and the brain region was disassociated. The nucleus such as periaqueductal gray (PAG), nucleus accumbens, striatum were selected for total RNA extraction. Differential display-PCR (DD-PCR) of mRNA was used for isolating the specific expressed genes. Three sets of anchor primers (T12MN, M=A,G,T or C; N=A,C or G) and 6 sets of arbitrary primers(AP1~AP6) were used for DDPCR amplification. By combination of anchor primers and arbitrary primers, series of PCR reaction were preformed. Results: More than 80 cDNA fragments, which might represent the differentially expressed genes, were obtained by DDPCR method. Dot blot was used for further selection of positive cDNA, and more than 10 cDNA fragments with strong positive signals were cloned. Four clones were sequenced. Four cDNA fragments were obtained. Conclusion: Based on the sequence homologous comparison with NIH BLAST data, four specially expressed new gene fragments have been isolated and cloned by using mRNA differential display-PCR (DDPCR) method.
, 百拇医药
(J Beijing Med Unvi, 1999,31:216-219)
mRNA差异显示技术(differential display PCR,DDPCR)是1992年由Liang等[1]建立,在筛选差异表达基因及新基因分离方面发挥了重要的作用[2,3]。在神经科学领域,DDPCR方法也有广泛的应用[4]。药物依赖是一个重要的研究课题,近年的基础研究业已发现一些重要的基因,如fos、jun、多巴胺受体及creb等基因在药物依赖的过程中起作用[5],但对药物成瘾的机制还远未明了。应用DDPCR的方法,直接从基因表达水平寻找并获得差异表达基因,进行功能分析,探讨其在药物依赖过程中的作用,将是一个有意义的研究方向。
1 材料与方法
1.1 材料
, 百拇医药
SD大鼠,雄性,250g,2月龄,购自北京医科大学实验动物中心;RNA提取试剂,购自GIBCO公司;DDPCR引物由Sybesyn公司合成,锚定引物(3组):,T12MN(M=A,T,G,C;N=A,C,G),随机引物(6组):AP1~AP6;TaqDNA聚合酶,逆转录酶(MMLV-RT),E.coliDNA聚合酶,E.coliDNA连接酶,T4DNA连接酶,购自BoehringerMannheim;RNasin,购自Promega;mRNA纯化试剂盒,购自Phamacia;cDNA合成试剂盒,cDNA消减杂交试剂盒,cDNA克隆试剂盒,购自Clontech;α-p32-dATP,购自北京亚辉生物医学公司。
1.2 实验大鼠的处理
大鼠分为实验组和对照组。实验组大鼠皮下注射递增剂量的吗啡:第1天5mg.kg-1,第2天10mg.kg-1,第3、4天20mg.kg-1,第5、6天40mg.kg-1,第7、8天80mg.kg-1,第9、10天120mg.kg-1;每天2次,连续注射7~10d,直至出现依赖症状。对照组大鼠注射同等剂量的生理盐水。最后一次注射后4h将大鼠断头,开颅,迅速切取大鼠的中脑导水管周围灰质(PAG)、伏核、纹状体,用于总RNA提取。
, 百拇医药
1.3 总RNA的提取
参考硫氰酸胍一步法稍加改进,取1mlRNA提取试剂,加入玻璃匀浆器中,将大鼠的脑组织置于匀浆器中,制备匀浆,转入1.5ml小离心管中,加100μl氯仿,于4℃,15000r.min-1,离心15min,取上清水相,转入新的离心管中,加等体积异丙醇沉淀RNA。RNA重溶于50μl水中,加10单位的RNasin,取2μlRNA样品进行琼脂糖凝胶电泳,检测RNA的完整性,取1μl进行紫外分光光度法检测RNA的浓度和纯度。其余样品-80℃保存。
1.4 逆转录反应
取3μg总RNA样品,加5单位无RNA酶活性的DNaseI消化样品中残存的DNA,在10μl体积下37℃保温20min,65℃保温5min,灭活DNaseI活性。加5单位RNasin,4μl5×1stBuffer,1μl10mmol.L-1dNTPs,1μloligodT锚定引物(T12G,或T12A,或T12C),5单位MMLV-RT逆转录酶,加水至20μl,37℃保温1h,65℃保温5min,灭活逆转录酶活性,样品可于-20℃存放。
, 百拇医药
1.5 PCR扩增
用3套锚定引物与6种(AP1~AP6)随机引物作不同组合,进行18个PCR反应,PCR反应所用的锚定引物应与逆转录的锚定引物相同。依次加入下列样品:逆转录产物2μl,10×PCRBuffer2.5μl,25mmol.L-1MgCl21.5μl,随机引物1μl,锚定引物1μl,10mmol.L-1dNTPs1 μl,TaqDNA聚合酶1μl,加水至25μl,滴加30μl石蜡油,按下列条件进行PCR扩增:94℃2min;94℃40s,40℃50s,72℃1.5min,40个循环;72℃7min。PCR结束后,先用琼脂糖凝胶电泳检测PCR扩增的结果,若结果满意,用聚丙烯酰胺凝胶电泳分离差异条带。
1.6 聚丙烯酰胺凝胶电泳
, 百拇医药
配制80g.L-1含8mol.L-1尿素的聚丙烯酰胺变性凝胶,凝胶20cm×40cm,PCR产物加入适量的6×loadingbuffer,每一泳道加入约20μl样品,于800~1000V电压下电泳3~4h,上样时对照组和实验组加载于相邻泳道,以便于比较条带差异。
1.7 银染法显示差异条带
电泳结束后按下述条件进行银染。凝胶在100g.L-1醋酸中浸泡20min,水洗10min,于染色液(AgNO33g,12.2mol.L-1甲醛0.7ml,加水1L)中染色30min,水洗10s,于显色液(Na2CO330g,20g.L-1硫代硫酸钠200μl,12.2mol.L-1甲醛1.5ml,加水1L)中显色5~10min,显色后于100g.L-1醋酸中浸泡10min,保存凝胶、拍照、比较结果。
, 百拇医药
1.8 差异条带的第二次PCR扩增及克隆
切取差异条带,加入20μl水,70℃水浴20min,取1μl作模板用相同的引物在相同的条件下进行PCR扩增,低熔点琼脂糖凝胶电泳纯化回收目标条带。
1.9 差异序列的斑点杂交
将第二次PCR扩增片段加载于尼龙膜上与cDNA探针进行杂交,杂交条件参考分子克隆实验手册。探针的制备:取与DDPCR相同的RNA样品,经逆转录反应合成双链cDNA,纯化后用随机引物标记试剂盒标记探针,放射性同位素为α-p32-dCTP。选取阳性扩增片段克隆于ClontechTA载体中并进行序列分析。
1.10 序列分析及序列比较
将序列分析所获得的数据与GeneBank数据库资料进行比较。NCBI同源性比较的网址:WWW.ncbi.nlm.nih.gov。
, http://www.100md.com
2 结果
2.1 总RNA的提取
用RNA提取的标准程序提取了大鼠的PAG、伏核、纹状体等核团样本中的总RNA。大鼠伏核的总RNA电泳图谱见图1。所提取的RNA完整、未降解,符合实验要求,28S/18S的比值大于1.5,同时用紫外分光光度法对RNA的纯度及浓度进行了测定,其OD260/280的比值接近2.0。

1, morphine treatment group; 2,control group. 5S, 18 S, 28 S RNA represent rRNA of total RNA, The higher ratio of 28S, the better quality of total RNA and its mRNA.
, 百拇医药
图1 伏核总RNA样品的电泳检测
Figure 1 Electrophoresis detection of total RNA sample
of N. Accumbens
2.2 吗啡依赖大鼠mRNA差异显示结果
经多轮次18组PCR扩增反应,获得了40余条差示条带,按1.7的方法对这些差示条带进行了第二次PCR扩增。图2为mRNA差异显示的图谱之一,图3是第二次PCR扩增的图谱。为了进一步鉴定这些差示条带,对这些差示条带进行了斑点杂交,初步克隆了10个杂交阳性片段。斑点杂交结果见图4。

AP1~AP4, represents different arbitrary primers; C, control group; T, morphine treatment group; M,PCR marker.
 represents differentially expressed cDNA bands.
represents differentially expressed cDNA bands., 百拇医药
图2 银染法mRNA差异显示电泳图谱之一
Figure 2 A picture of the DDPCR amplification by silver staining
1~10 represent different cDNA fragment clones.

图3 差异表达产物的cDNA片段的第2轮PCR扩增
Figure 3 Second round PCR amplification
of differentially expressed cDNA fragments
, http://www.100md.com a, b, c, d represent cDNA fragments of pAC11, pAC12,pAC13, pAC14, respectively.

图4 斑点杂交结果
Figure 4 The result of dot blot
2.3 差示条带的序列分析及比较
对4个强阳性克隆进行了序列分析,并通过BLAST数据库进行了序列的同源性比较。比较结果显示这4个片段均为新基因片段,在数据库中查询未见同源序列。4个阳性片段的数据库查询部分结果是pAC11:Score=38, E Value=0.65; pAC12: Score=40, E Value=0.18; pAC13: Score=40。 E Value=0.24; pAC14: Score=40, E Value=0.54。Gene Bank登录情况为pAC11, AF145788; pAC12, AF145789; pAC13, AF145794; pAC14, AF145795, AF113006。图5列出了4个cDNA片段的序列。

, http://www.100md.com
图5 4个新cDNA片段的序列
Figure 5 The sequence of 4 novel cDNA fiagments
3 讨论
分离新基因的战略目前倍受重视,通过差异分析,获得在不同处理过程中的细胞或动物的特异表达的cDNA在目前是一个行之有效的方法,也比较符合我国国情。本研究利用DDPCR方法获得了4个差异表达产物的cDNA片段,通过Gene Bank数据库同源性比较,没有发现同源序列。下一步的主要工作是进行Northern杂交分析,预测全长的mRNA的长度;脑切片的原位杂交,分析其在脑内的表达情况;通过RACE扩增的方法,获得全长的cDNA,通过氨基酸序列推导预测其功能,并进行一系列的功能分析。
根据文献[6~8]报道及我们实验的体会,DDPCR技术虽然是行之有效的扩增新基因的方法,但也会产生大量的假阳性片段,这是一个难以克服的缺点。我们的解决办法是获得扩增片段后,先进行斑点杂交,标记cDNA探针进行同组杂交及交叉杂交(同组杂交:实验组探针同实验组片段杂交,对照组探针同对照组片段杂交;交叉杂交:实验组探针同对照组片段杂交;对照组片段同实验组片段杂交),来筛选较有意义的差异片段。对这些片段进行序列分析及比较,避免大规模测序造成的人力和财力的浪费。斑点杂交的方法有许多文献报道[9,10],我们的方法略有改进,这样更易于操作同时又能保证阳性结果。我们使用的探针是用作DDPCR相同的总RNA,通过逆转录反应合成cDNA的第一链和第二链,合成的cDNA基本上代表了样品中原有的mRNA序列,用随机引物方法标记探针,分别同获得的差异片段进行杂交,只挑选在同组杂交中阳性而交叉杂交阴性的片段作进一步分析。我们的实验结果证实这一方法是行之有效的,具有较强的可信度及可操作性,能有效地克服DDPCR的假阳性高、工作量大的缺点。我们同时还进行了用抑制性消减杂交分离吗啡依赖大鼠特异表达基因的研究工作,也取得了部分实验结果,将另文发表。
, 百拇医药
* 国家自然科学基金(39600192)资助项目。
参考文献
1 Liang P, Pardee AB. Differential display of eukaryotic messenger RNA by means of the polymerase chain reaction. Science, 1992, 257:967-971
2 Hsu DKW, Donohue PJ, Alberts GF, et al. Fibroblast growth factor-1 induces phosphofructokinase fatty acid synthase and Ca2+-ATPase mRNA expression in NIH3T3 cells. Biochem Biophys Res Comm, 1993, 197:1483-1491
, 百拇医药
3 McClelland M, Mathieu-Daude F, Welsh J. Fingerprinting and differential display using arbitrary primed PCR. Trends Genet, 1995, 11:242-246
4 Babity JM, Newton RA, Guido ME, et al. The application of differential display to the brain: adaptations for the study of heterogeneous tissue. In: Liang P, Pardee AB, ed. Methods in Molecular Biology. Iotowa:Humana Press, 1997 (V85).285-295
5 Nestler EJ. Molecular mechanisms underlying opiate addication: implications of for medication development. Neuroscience, 1997, 9:84-93
, 百拇医药
6 Bertioli DJ, Schlichter UH, Adams MJ, et al. An analysis of differential display shows a strong bias towards high copy number mRNAs. Nucl Acids Res, 1995, 23:4520-4523
7 Debouck C. Differential display or differential dismay? Curr Opin Biotechnol, 1995, 6:597-599
8 Wadhwa R, Duncan E, Kaul SC, et al. An effective elimination of false positives isolated from differential display of mRNAs. Mol Biotechnol, 1996, 6:213-217
, 百拇医药
9 Liang P, Averboukh L, Pardee AB. Distribution and cloning of eukaryotic mRNAs by means of differential display: refinements and optimization. Nucl Acids Res, 1993, 21:3269-3275
10 Poirier GMC, Pyati J, Wan JS, et al. Screening differentially expressed cDNA clones obtained by differential display using amplified RNA. Nucl Acids Res, 1997, 25:913-914
(1999-03-09收稿), 百拇医药