玻璃珠-盐析法提取常见致病真菌DNA的研究
http://www.100md.com
中华皮肤科杂志 2000年第5期第33卷
作者:秦振宇 吴绍熙 Roy L.Hopfer 吕桂霞 姚聪君 郭宁如
单位:吴绍熙(210042南京,中国医学科学院 中国协和医科大学皮肤病研究所指导者);吕桂霞 秦振宇 郭宁如(210042南京,中国医学科学院 中国协和医科大学皮肤病研究所);Roy L. Hopfer(UNC Hospitals, University of North Carolina)
关键词:真菌;致病性;DNA;提取
中华皮肤科杂志000510
【摘要】目的进一步提高提取DNA的效率并减少污染和毒性,以适应临床PCR检测的需要,建立玻璃珠-盐析法快速提取常见致病真菌DNA。方法分别采用玻璃珠-盐析法和经典的酶解-酚氯仿法比较从白念珠菌、乳酒念珠菌、克柔念珠菌、季也蒙念珠菌、新生隐球菌、烟曲霉、黄曲霉和马内菲青霉等8种常见致病真菌中提取DNA,同时行PCR以评价其可靠性。结果本方法从8种常见致病真菌均提取浓度较高且较完整的DNA,用于PCR也获得良好结果。结论玻璃珠-盐析法提取致病酵母菌和丝状真菌安全、快速且有效。
, 百拇医药
Glass Beads- Salting out Procedure for Extracting DNA from Common Pathogenic Fungi
QIN Zhenyu,WU Shaoxi,Roy L.Hopfer, et al.
(Institute of Dermatology, Chinese Academy of Medical Sciences, Peking Union Medical College, Nangjing 210042)
【Abstract】 Objective To develop glass- beads and salting- out procedure for extracting DNA from common pathogenic fungi. Methods Glass beads- salting out procedure was used to isolated DNA from 8 pathogenic fungi, including Candida albicans, Candida kefyr, Candida krusei, Candida guilliermondii, Cryptococcus neoformans, Aspergillus fumigatus, Aspergillus flavus and Penicillium marneffei, and compared with conventional phenol- chloroform extraction method. The efficiency of this procedure was estimated by using PCR. Results For all the eight organisms, the rapid extraction procedure resulted in high yield and intact DNA. Conclusion The disruption of fungal cells by glass beads- salting out procedure is a safe, rapid, and efficient method for extraction of genomic DNA from medically important yeasts and filamentous fungi.
, 百拇医药
【Key words】 Fungi, pathogenic DNA Extraction
聚合酶链反应(PCR)技术在医学真菌临床上的应用,一是用于早期诊断[1],二是用于菌株分型以确定其相似性,进而明确感染来源[2]。为了保证PCR的敏感性和特异性,要求所提取的DNA纯度较高,不含抑制PCR反应的成分,同时也应快速和易于操作以满足临床的需要。玻璃珠法是一种以直径0.45~0.50mm玻璃珠振荡机械性地破坏真菌细胞壁的DNA提取方法,目前研究发现其破坏真菌壁的效率最高。盐析法是一种较新的通过饱和氯化钠纯化DNA的有效技术,已广泛应用于血细胞的提取之中。我们在实际工作中将两者相结合建立玻璃珠-盐析法(GS),发现提取效率较佳。为进一步研究这一技术,我们将其与传统的酶解-酚氯仿抽提法(LC)进行比较,现报道如下。
材料和方法
一、菌种
, 百拇医药
白念珠菌(IDC1a)、乳酒念珠菌(J941267)、克柔念珠菌(B46120)、季也蒙念珠菌(ID177)、新生隐球菌(ID44)、烟曲霉(ID203)、黄曲霉(ID204)和马内菲青霉(ID260)各1株。真菌菌种来源于中国微生物菌种保藏管理委员会医学真菌中心,均为标准菌株。
二、试剂与设备
玻璃珠为B.BraunMelsungenAG产品,TaqDNA聚合酶系北京大学免疫学教研室产品,dNTPs为SABC产品。高速振荡器根据旋涡混合器(QL-901型,江苏海门其林医用仪器厂)改装,在座托上加双层海绵后再压以重物,旁边设支架支撑以保持振荡时平衡。
三、菌种的培养和收集[3]
酵母菌在沙堡培养基培养24h后,取单个菌落(直径2mm)接种于5mLYPD液基中,念珠菌属30℃,350r/min下振荡培养24h,隐球菌属27℃,350r/min下振荡培养48h。2000×g离心10min集菌,以生理盐水洗涤1次。曲霉和青霉在PDA培养基30℃培养24h后,转到室温下至成熟。用生理盐水冲洗菌落以获得分生孢子。2000×g离心10min集菌,以生理盐水洗涤1次。以血细胞计数器将每一菌种浓度调至108细胞/mL,再离心后弃上清液,至1.5mL微量离心管中,-20℃备用。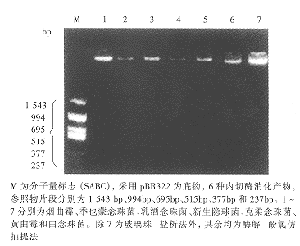
, 百拇医药
图1部分真菌DNA在质量分数为0.8%琼脂糖凝胶电泳结果
四、玻璃珠-盐析法提取DNA
加入300μL提取液[2%TritonX-100,1%SDS,100mmol/LNaCl,10mmol/LTris-Cl(pH8.0),1mmol/LEDTA]悬浮菌体,再加入300mg玻璃珠,在高速振荡器上振荡,酵母菌为5min,曲霉和青霉为30min。200μL预冷饱和NaCl(约为6mol/L),用力振荡15s,再以2500r/min离心15min。取上清液转移到另一离心管中,加入2倍体积的无水乙醇上下颠倒,离心10min,弃上清液后干燥。最后,加入100μL10mmol/LTris(pH8.0),1mmol/LEDTA和1μLRNase(SABC),37℃孵育1h。-20℃保存。
五、酶解-酚氯仿抽提法提取DNA
, 百拇医药
加入100μL山梨醇溶液[0.9mol/L山梨醇,0.1mol/LTris(pH7.6),0.1mmol/LEDTA]悬浮菌体,再加入50μL0.3mg/mL的溶细胞酶(以山梨醇溶液配制)和50μL0.28mol/L2-巯基乙醇,混合后37℃孵育3h。再加入裂解缓冲液50μL[0.05mol/LEDTA(pH8.0),0.3%SDS]65℃孵育30min,酚/氯仿/异戊醇抽提直到界面不再有白色物质。取上清液转移到另一离心管中,加入2倍体积的无水乙醇上下颠倒,离心10min,弃上清液后干燥。最后,加入100μL10mmol/LTris(pH8.0),1mmol/LEDTA和1μLRNase(SABC),37℃孵育1h。-20℃保存。
六、PCR
参照文献[3]。
七、提取DNA浓度检测
, 百拇医药
提取的DNA在质量分数为0.8%琼脂糖凝胶电泳、溴化乙锭染色,中波紫外线观察、照相,并与DNA标准物比较。采用1D软件(SELEquipmentCo.Ltd)获得电泳条带的绝对A值,再以SAS统计软件(6.02)行线性回归分析,绘出标准曲线,并据此推算出提取DNA的含量。
八、统计学方法
两种DNA提取方法各重复3次,两组数据做Wilcoxon秩和检验。
结果
一、提取的DNA含量
酚氯仿抽提法和盐析法所得的DNA在质量分数为0.8%琼脂糖凝胶电泳上均为清晰完整的条带(见图1)。所得的DNA含量见表1。对两组数据作Wilcoxon秩和检验,P=0.0239,<0.05,表明这两种方法提取的DNA含量差异有显著性。
, 百拇医药
二、PCR结果
两组DNA提取方法均产生310bp特异性条带。
表1玻璃珠-盐析法和酶解-酚氯仿抽提法提取DNA浓度比较( ±s)μg/mL
±s)μg/mL
菌株
玻璃珠-盐析法
酶解-酚氯仿抽提法
白念珠菌
15.25±1.24
10.24±0.67
乳酒念珠菌
, 百拇医药
12.76±0.98
8.56±0.95
克柔念珠菌
14.34±1.03
9.74±0.76
季也蒙念珠菌
11.28±0.82
8.26±0.36
新生隐球菌
4.36±0.31
5.73±0.14
烟曲霉
, http://www.100md.com
15.56±1.34
11.26±0.89
黄曲霉
14.76±0.99
12.73±1.02
马内菲青霉
11.72±0.79
10.71±0.91
讨论
致病真菌DNA的提取方法种类繁多,同时又不断有新的方法问世如CTAB法[4]和硅化膜法。玻璃珠/酚、酶解酶/蛋白酶K/硅化膜和蛋白酶K/硅化膜3种方法效果较好,并且第1种所需时间最短,第2种最简便,第3种敏感性最高。需要说明,在我国已有使用酶解酶的文献报道,该酶和本实验中的溶细胞酶同为β-1,3-葡聚糖酶产品,后者水解真菌中β-1,3-葡聚糖连接链中的葡萄糖多聚体致细胞壁部分缺失,增加对渗透压的敏感性。但酶解酶是一种自然的β-1,3-葡聚糖酶,为Arthrobacterluteus水中培养后的发酵产物,而溶细胞酶是一种合成的β-1,3-葡聚糖酶等同物。溶细胞酶可避免酶解酶中不纯物质如蛋白酶、苷露糖酶、淀粉酶、木糖酶、磷酸酶和微量的DNA酶。Loffler等[5]对5种常用试剂盒(QIAmpTissue、基因释放剂、PuregeneD600、Dynabeads和DNAzol)提取血中致病真菌DNA方法比较,其敏感性在10~103细胞/mL之间,所需时间为3.25~4h,价格0.80~2.30美元/样本。其中QIAmpTissue的敏感性和纯度最佳。在我国已有使用基因释放剂的文献报道,但我们的经验和国外报道均表明其提取真菌DNA纯度不佳而可能抑制DNA聚合,导致低敏感性或假阴性结果。
, 百拇医药
在Haugland等[6]的研究中,以定量竞争PCR的方法评价液氮、室温下研磨、超声、冻融和玻璃珠法所提取的杂色曲霉等5种真菌的DNA。结果,玻璃珠法在所提取DNA的绝对拷贝数和最小提取变异两项指标的效率最高。通过计算每一个分生孢子的平均模板数发现玻璃珠法所提取的DNA足够PCR检测10个分生孢子。在vanBurik等[7]的研究中,比较玻璃珠旋转法、研磨后玻璃珠粉碎法、1%十六三乙基溴化胺(CTAB)缓冲液加入玻璃珠后水浴超声粉碎、CTAB中水浴超声粉碎、研磨后CTAB中孵育和溶细胞酶酶解法等6种方法提取烟曲霉的效率。以分光光度法评价纯度,琼脂糖电泳法评价DNA被剪切的情况。结果综合评价,玻璃珠旋转法最好,其次为研磨后CTAB中孵育=研磨后玻璃珠粉碎法>1%CTAB缓冲液加入玻璃珠后水浴超声粉碎=CTAB中水浴超声粉碎=溶细胞酶酶解法。但是,玻璃珠法一般要采用酚氯仿抽提DNA。酚氯仿抽提法是目前最常用的方法之一,所获的DNA纯度高,可满足各种检测技术的要求,但该法操作重蒸饱和酚的处理复杂,有污染和毒性作用。Miller等[8]和谭建明等[9]等建立盐析法以替代酚氯仿抽提,操作简单,所需试剂易得,无污染和有害作用。本实验用准确度较高的DNA标准物对照法,将其与玻璃珠法联合,提取浓度高于传统的酶解法。
, 百拇医药
志谢西安医科大学计算机中心陈康高级工程师协助图像分析
卫生部临床医学重点课题
参考文献
[1]Yamakami Y, et al. Evaluation of PCR for detection of DNA specific for Aspergillus species in sera of patients with various forms of pulmonary aspergillosis. J Clin Microbiol, 1998,36:3619- 3623.
[2]Okhravi N, Adamson P, Mant R, et al. Polymerase chain reaction and restriction fragment length polymorphism mediated detection and specification of Candida spp. causing intraocular infection. Invest Ophthalmol Vis Sci, 1998,39:859- 866.
, 百拇医药
[3]Muller FC, Werner KE, Kasai M, et al. Rapid extraction of genomic DNA from medically important yeasts and filamentous fungi by high- speed cell disruption. J Clin Microbiol, 1998, 36:1625- 1629.
[4]Velegraki A, Kambouris M, Kostourou A, et al. Rapid extraction of fungal DNA from clinical samples for PCR amplification. Med Mycol, 1999,37:69- 73.
[5]Loffler J, Hebart H, Schumacher U, et al. Comparison of different methods for extraction of DNA of fungal pathogens from cultures and blood. J Clin Microbiol, 1997,35:3311- 3312.
, 百拇医药
[6]Haugland RA, Heckman JL, Wymer LJ. Evaluation of different methods for the extraction of DNA from fungal conidia by quantitative competitive PCR analysis. J Microbiol Methods, 1999,37:165- 176.
[7]van Burik JAH, Schreckhise RW, White TC, et al. Comparison of six extraction techniques for isolation of DNA from filamentous fungi. Med Mycol, 1998,36:299- 303.
[8]Miller S, Dykes D, Polesky H. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res, 1988,16:1215.
[9]谭建明,谢桐,徐琴君,等.快速盐析法提取DNA在HLA基因分型中的应用.中华器官移植杂志,1996,17:9-11.
(收稿日期:1999-10-14), http://www.100md.com
单位:吴绍熙(210042南京,中国医学科学院 中国协和医科大学皮肤病研究所指导者);吕桂霞 秦振宇 郭宁如(210042南京,中国医学科学院 中国协和医科大学皮肤病研究所);Roy L. Hopfer(UNC Hospitals, University of North Carolina)
关键词:真菌;致病性;DNA;提取
中华皮肤科杂志000510
【摘要】目的进一步提高提取DNA的效率并减少污染和毒性,以适应临床PCR检测的需要,建立玻璃珠-盐析法快速提取常见致病真菌DNA。方法分别采用玻璃珠-盐析法和经典的酶解-酚氯仿法比较从白念珠菌、乳酒念珠菌、克柔念珠菌、季也蒙念珠菌、新生隐球菌、烟曲霉、黄曲霉和马内菲青霉等8种常见致病真菌中提取DNA,同时行PCR以评价其可靠性。结果本方法从8种常见致病真菌均提取浓度较高且较完整的DNA,用于PCR也获得良好结果。结论玻璃珠-盐析法提取致病酵母菌和丝状真菌安全、快速且有效。
, 百拇医药
Glass Beads- Salting out Procedure for Extracting DNA from Common Pathogenic Fungi
QIN Zhenyu,WU Shaoxi,Roy L.Hopfer, et al.
(Institute of Dermatology, Chinese Academy of Medical Sciences, Peking Union Medical College, Nangjing 210042)
【Abstract】 Objective To develop glass- beads and salting- out procedure for extracting DNA from common pathogenic fungi. Methods Glass beads- salting out procedure was used to isolated DNA from 8 pathogenic fungi, including Candida albicans, Candida kefyr, Candida krusei, Candida guilliermondii, Cryptococcus neoformans, Aspergillus fumigatus, Aspergillus flavus and Penicillium marneffei, and compared with conventional phenol- chloroform extraction method. The efficiency of this procedure was estimated by using PCR. Results For all the eight organisms, the rapid extraction procedure resulted in high yield and intact DNA. Conclusion The disruption of fungal cells by glass beads- salting out procedure is a safe, rapid, and efficient method for extraction of genomic DNA from medically important yeasts and filamentous fungi.
, 百拇医药
【Key words】 Fungi, pathogenic DNA Extraction
聚合酶链反应(PCR)技术在医学真菌临床上的应用,一是用于早期诊断[1],二是用于菌株分型以确定其相似性,进而明确感染来源[2]。为了保证PCR的敏感性和特异性,要求所提取的DNA纯度较高,不含抑制PCR反应的成分,同时也应快速和易于操作以满足临床的需要。玻璃珠法是一种以直径0.45~0.50mm玻璃珠振荡机械性地破坏真菌细胞壁的DNA提取方法,目前研究发现其破坏真菌壁的效率最高。盐析法是一种较新的通过饱和氯化钠纯化DNA的有效技术,已广泛应用于血细胞的提取之中。我们在实际工作中将两者相结合建立玻璃珠-盐析法(GS),发现提取效率较佳。为进一步研究这一技术,我们将其与传统的酶解-酚氯仿抽提法(LC)进行比较,现报道如下。
材料和方法
一、菌种
, 百拇医药
白念珠菌(IDC1a)、乳酒念珠菌(J941267)、克柔念珠菌(B46120)、季也蒙念珠菌(ID177)、新生隐球菌(ID44)、烟曲霉(ID203)、黄曲霉(ID204)和马内菲青霉(ID260)各1株。真菌菌种来源于中国微生物菌种保藏管理委员会医学真菌中心,均为标准菌株。
二、试剂与设备
玻璃珠为B.BraunMelsungenAG产品,TaqDNA聚合酶系北京大学免疫学教研室产品,dNTPs为SABC产品。高速振荡器根据旋涡混合器(QL-901型,江苏海门其林医用仪器厂)改装,在座托上加双层海绵后再压以重物,旁边设支架支撑以保持振荡时平衡。
三、菌种的培养和收集[3]
酵母菌在沙堡培养基培养24h后,取单个菌落(直径2mm)接种于5mLYPD液基中,念珠菌属30℃,350r/min下振荡培养24h,隐球菌属27℃,350r/min下振荡培养48h。2000×g离心10min集菌,以生理盐水洗涤1次。曲霉和青霉在PDA培养基30℃培养24h后,转到室温下至成熟。用生理盐水冲洗菌落以获得分生孢子。2000×g离心10min集菌,以生理盐水洗涤1次。以血细胞计数器将每一菌种浓度调至108细胞/mL,再离心后弃上清液,至1.5mL微量离心管中,-20℃备用。

, 百拇医药
图1部分真菌DNA在质量分数为0.8%琼脂糖凝胶电泳结果
四、玻璃珠-盐析法提取DNA
加入300μL提取液[2%TritonX-100,1%SDS,100mmol/LNaCl,10mmol/LTris-Cl(pH8.0),1mmol/LEDTA]悬浮菌体,再加入300mg玻璃珠,在高速振荡器上振荡,酵母菌为5min,曲霉和青霉为30min。200μL预冷饱和NaCl(约为6mol/L),用力振荡15s,再以2500r/min离心15min。取上清液转移到另一离心管中,加入2倍体积的无水乙醇上下颠倒,离心10min,弃上清液后干燥。最后,加入100μL10mmol/LTris(pH8.0),1mmol/LEDTA和1μLRNase(SABC),37℃孵育1h。-20℃保存。
五、酶解-酚氯仿抽提法提取DNA
, 百拇医药
加入100μL山梨醇溶液[0.9mol/L山梨醇,0.1mol/LTris(pH7.6),0.1mmol/LEDTA]悬浮菌体,再加入50μL0.3mg/mL的溶细胞酶(以山梨醇溶液配制)和50μL0.28mol/L2-巯基乙醇,混合后37℃孵育3h。再加入裂解缓冲液50μL[0.05mol/LEDTA(pH8.0),0.3%SDS]65℃孵育30min,酚/氯仿/异戊醇抽提直到界面不再有白色物质。取上清液转移到另一离心管中,加入2倍体积的无水乙醇上下颠倒,离心10min,弃上清液后干燥。最后,加入100μL10mmol/LTris(pH8.0),1mmol/LEDTA和1μLRNase(SABC),37℃孵育1h。-20℃保存。
六、PCR
参照文献[3]。
七、提取DNA浓度检测
, 百拇医药
提取的DNA在质量分数为0.8%琼脂糖凝胶电泳、溴化乙锭染色,中波紫外线观察、照相,并与DNA标准物比较。采用1D软件(SELEquipmentCo.Ltd)获得电泳条带的绝对A值,再以SAS统计软件(6.02)行线性回归分析,绘出标准曲线,并据此推算出提取DNA的含量。
八、统计学方法
两种DNA提取方法各重复3次,两组数据做Wilcoxon秩和检验。
结果
一、提取的DNA含量
酚氯仿抽提法和盐析法所得的DNA在质量分数为0.8%琼脂糖凝胶电泳上均为清晰完整的条带(见图1)。所得的DNA含量见表1。对两组数据作Wilcoxon秩和检验,P=0.0239,<0.05,表明这两种方法提取的DNA含量差异有显著性。
, 百拇医药
二、PCR结果
两组DNA提取方法均产生310bp特异性条带。
表1玻璃珠-盐析法和酶解-酚氯仿抽提法提取DNA浓度比较(
 ±s)μg/mL
±s)μg/mL菌株
玻璃珠-盐析法
酶解-酚氯仿抽提法
白念珠菌
15.25±1.24
10.24±0.67
乳酒念珠菌
, 百拇医药
12.76±0.98
8.56±0.95
克柔念珠菌
14.34±1.03
9.74±0.76
季也蒙念珠菌
11.28±0.82
8.26±0.36
新生隐球菌
4.36±0.31
5.73±0.14
烟曲霉
, http://www.100md.com
15.56±1.34
11.26±0.89
黄曲霉
14.76±0.99
12.73±1.02
马内菲青霉
11.72±0.79
10.71±0.91
讨论
致病真菌DNA的提取方法种类繁多,同时又不断有新的方法问世如CTAB法[4]和硅化膜法。玻璃珠/酚、酶解酶/蛋白酶K/硅化膜和蛋白酶K/硅化膜3种方法效果较好,并且第1种所需时间最短,第2种最简便,第3种敏感性最高。需要说明,在我国已有使用酶解酶的文献报道,该酶和本实验中的溶细胞酶同为β-1,3-葡聚糖酶产品,后者水解真菌中β-1,3-葡聚糖连接链中的葡萄糖多聚体致细胞壁部分缺失,增加对渗透压的敏感性。但酶解酶是一种自然的β-1,3-葡聚糖酶,为Arthrobacterluteus水中培养后的发酵产物,而溶细胞酶是一种合成的β-1,3-葡聚糖酶等同物。溶细胞酶可避免酶解酶中不纯物质如蛋白酶、苷露糖酶、淀粉酶、木糖酶、磷酸酶和微量的DNA酶。Loffler等[5]对5种常用试剂盒(QIAmpTissue、基因释放剂、PuregeneD600、Dynabeads和DNAzol)提取血中致病真菌DNA方法比较,其敏感性在10~103细胞/mL之间,所需时间为3.25~4h,价格0.80~2.30美元/样本。其中QIAmpTissue的敏感性和纯度最佳。在我国已有使用基因释放剂的文献报道,但我们的经验和国外报道均表明其提取真菌DNA纯度不佳而可能抑制DNA聚合,导致低敏感性或假阴性结果。
, 百拇医药
在Haugland等[6]的研究中,以定量竞争PCR的方法评价液氮、室温下研磨、超声、冻融和玻璃珠法所提取的杂色曲霉等5种真菌的DNA。结果,玻璃珠法在所提取DNA的绝对拷贝数和最小提取变异两项指标的效率最高。通过计算每一个分生孢子的平均模板数发现玻璃珠法所提取的DNA足够PCR检测10个分生孢子。在vanBurik等[7]的研究中,比较玻璃珠旋转法、研磨后玻璃珠粉碎法、1%十六三乙基溴化胺(CTAB)缓冲液加入玻璃珠后水浴超声粉碎、CTAB中水浴超声粉碎、研磨后CTAB中孵育和溶细胞酶酶解法等6种方法提取烟曲霉的效率。以分光光度法评价纯度,琼脂糖电泳法评价DNA被剪切的情况。结果综合评价,玻璃珠旋转法最好,其次为研磨后CTAB中孵育=研磨后玻璃珠粉碎法>1%CTAB缓冲液加入玻璃珠后水浴超声粉碎=CTAB中水浴超声粉碎=溶细胞酶酶解法。但是,玻璃珠法一般要采用酚氯仿抽提DNA。酚氯仿抽提法是目前最常用的方法之一,所获的DNA纯度高,可满足各种检测技术的要求,但该法操作重蒸饱和酚的处理复杂,有污染和毒性作用。Miller等[8]和谭建明等[9]等建立盐析法以替代酚氯仿抽提,操作简单,所需试剂易得,无污染和有害作用。本实验用准确度较高的DNA标准物对照法,将其与玻璃珠法联合,提取浓度高于传统的酶解法。
, 百拇医药
志谢西安医科大学计算机中心陈康高级工程师协助图像分析
卫生部临床医学重点课题
参考文献
[1]Yamakami Y, et al. Evaluation of PCR for detection of DNA specific for Aspergillus species in sera of patients with various forms of pulmonary aspergillosis. J Clin Microbiol, 1998,36:3619- 3623.
[2]Okhravi N, Adamson P, Mant R, et al. Polymerase chain reaction and restriction fragment length polymorphism mediated detection and specification of Candida spp. causing intraocular infection. Invest Ophthalmol Vis Sci, 1998,39:859- 866.
, 百拇医药
[3]Muller FC, Werner KE, Kasai M, et al. Rapid extraction of genomic DNA from medically important yeasts and filamentous fungi by high- speed cell disruption. J Clin Microbiol, 1998, 36:1625- 1629.
[4]Velegraki A, Kambouris M, Kostourou A, et al. Rapid extraction of fungal DNA from clinical samples for PCR amplification. Med Mycol, 1999,37:69- 73.
[5]Loffler J, Hebart H, Schumacher U, et al. Comparison of different methods for extraction of DNA of fungal pathogens from cultures and blood. J Clin Microbiol, 1997,35:3311- 3312.
, 百拇医药
[6]Haugland RA, Heckman JL, Wymer LJ. Evaluation of different methods for the extraction of DNA from fungal conidia by quantitative competitive PCR analysis. J Microbiol Methods, 1999,37:165- 176.
[7]van Burik JAH, Schreckhise RW, White TC, et al. Comparison of six extraction techniques for isolation of DNA from filamentous fungi. Med Mycol, 1998,36:299- 303.
[8]Miller S, Dykes D, Polesky H. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res, 1988,16:1215.
[9]谭建明,谢桐,徐琴君,等.快速盐析法提取DNA在HLA基因分型中的应用.中华器官移植杂志,1996,17:9-11.
(收稿日期:1999-10-14), http://www.100md.com