甄氏外瓶霉的PCR-RFLP分型研究
http://www.100md.com
中华皮肤科杂志 2000年第5期第33卷
作者:李东明 李若瑜 王端礼 王晓红 马圣清
单位:100034 北京大学第一医院皮肤科
关键词:甄氏外瓶霉;随机扩增多态DNA技术
中华皮肤科杂志000503
【摘要】目的对不同来源的甄氏外瓶霉进行分析,为建立更为合理的分型方法提供依据。方法以ITS3和ITS4为引物,对17株甄氏外瓶霉的核糖体DNA转录间隔区(ITSⅡ)进行扩增,4种内切酶(HinFⅠ、MspⅠ、BsuRⅠ和RsaⅠ)酶切。结果ITSⅡ区扩增片段约为330bp,17个菌株可分为Ⅵ型,3株变种分属于不同的型,BMU00195带型独特,有可能为新变种或新种;种群关系与来源显示出一定的相关性。结论PCR-RFLP用于甄氏外瓶霉的分型研究较为可靠,对于确定有争议菌种的分类学地位具有重要的意义。
, http://www.100md.com
PCR-ribotyping of Exophiala jeanselmei
LI Dongming,LI Ruoyu,WANG Duanli,et al.
(Department of Dermatology, First Hospital, Peking University, Beijing 100034)
【Abstract】 Objective To analyze Exophiala jeanselmei strains recovered from different sources and to establish a better method for typing the strains in this species. Methods The ribosomal internal transcribed spacer regions (ITSⅡ ) of 17 isolates Exophiala jeanselmei were amplified with primer ITS3 and ITS4 and digested with four restriction enzymes (HinFⅠ , MspⅠ , BsuRⅠ and RsaⅠ ). Results The amplicon length of the ITSⅡ was about 330bp, and 6 types of characteristic patterns for the species were found after restriction analysis. The variants of E. jeanselmei were unrelated and should therefore be maintained as separate species. The unique patterns of isolate BMU00195 were shown,which seems probably to be a new variant or even a new species. Conclusions PCR- RFLP is useful for the typing and identification of Exophiala jeanselmei, which is important in confirming the taxonomic position of a complex species.
, 百拇医药
【Key words】 Exophiala jeanselmei Random amplified polymorphic DNA technique
外瓶霉(Exophialaspp,E)为暗色丝孢霉病的主要致病菌之一,而甄氏外瓶霉(E.jeanselmei)是外瓶霉属中致病性较强的一个种,易侵犯皮肤、上消化道及心血管系统,且病灶可血行播散[1,2]。该种内各亚种间变异性较大,且侵袭力、毒力均有差别,对药物的反应性亦不同。由于该类真菌形态及生理生化性质差异较小,传统的分型方法往往不能满足需要,分子生物学技术的发展为从基因水平上揭示真菌的亲缘关系提供了新的手段。近年来,核糖体DNA(rDNA)内转录间隔区(internaltranscribedspacer,ITS)已被应用于外瓶霉及其相近属种的分类中。本实验在国际上首次将PCR-RFLP应用于甄氏外瓶霉的种内分型研究,以期为建立更为合理的分类体系提供依据。
材料和方法
, 百拇医药
一、标本来源
包括甄氏外瓶霉及其变种共17株,均为北京大学真菌和真菌病研究中心保存菌株。
二、实验方法
————————
(一)DNA提取:将实验菌株接种于PDA,27℃培养2周,收集菌丝体约0.1g,用氯化苄法提取DNA[3],并在质量分数为0.8%琼脂糖凝胶上检测DNA的量和浓度。
(二)PCR扩增:应用引物ITS3和ITS4[4](华美公司合成)对ITSⅡ进行扩增。50μL反应体系含10mmolTris·HCl,1.5mmolMgCl,800μmol4dNTP,0.4μmolITS3,0.4μmolITS4TaqDNA聚合酶1U。反应条件:94℃变性1min,52℃退火50s,72℃延伸1min20s,35个循环。
, 百拇医药
(三)酶切:将PCR产物分别加入MspⅠ、BsuRⅠ、RsaⅠ和HinFⅠ37℃孵育3h,纯化后用质量分数为2%琼脂糖凝胶分离,以puc19DNA经MspⅠ彻底水解后的产物(MBI公司生产)作为分子量标记。紫外灯下照相,并在UV凝胶成象分析系统中成象。
(四)统计分析:应用单匹配系数(Singlematchingcoefficient,SM)表示物种的亲缘关系,计算方法为:SM=a/(a+b),式中a为两菌共有的“1”和“0”的条带数,“b”为非共有的条带数。菌株间的相似性为:SM×%。将每个菌的“1”和“0”输入,经Ntsys软件分析得到单匹配系数矩阵图,用该软件的UPGMA聚类分析法生成表示菌株间亲缘关系的树状图。
图1甄氏外瓶霉ITSⅡ区酶切带型图
结果
, http://www.100md.com
各菌株ITS间区扩增片段约为330bp,4种内切酶酶切后带型见表1。HinFI只对北京大学真菌和真菌病研究中心(BMU)00195(E.jeanselme ivar.lecanii-corni)有一个识别位点,MapⅠ及RsaⅠ对多数菌株均具有一个以上的切点,BsuRⅠ则只对半数以上菌株的ITS区有识别位点(图1)。
多种内切酶酶切带型综合聚类分析见图2,树状图主要分为6支,第1支包括模式株在内共6株菌,其中模式株BMU00001与标准株BMU00282、BMU00481、BMU00457及BMU00011、BMU00003带型完全相同(SM=1),与BMU00007相似程度亦较高(SM=0.875);第2支中3株菌为自然分离株(SM=1);第3支中分离自患者的BMU00007及BMU00012显示出高度相似性(SM=1);在第4支中,临床分离株BMU00014与BMU00010(E.jeanselme ivar.heteromorpha)相似程度较高(SM=0.875);分离自患者的BMU00195(E.jeanselme ivar.lecanii-corni)带型独特,单独成为第5支。第6支包括2株环境分离株和1株E.jeanselme ivar.lecanii-corni(BMU00480)。3株变种间相似程度较低(SM<0.51),而E.jeanselme ivar.heteromorph(BMU00457)却与标准菌株之间存在高度相似性。
, http://www.100md.com
表1甄氏外瓶霉ITSⅡ间区酶切带型表
菌号(BMU)
HinFⅠ
MspⅠ
BsuRⅠ
RsaⅠ
00001
A
A
A
A
00011
A
, 百拇医药
A
A
B
00003
A
A
A
A
00007
A
A
A
C
00006
, 百拇医药
A
B
B
A
00004
A
B
B
A
00002
A
A
B
A
, 百拇医药
00009
A
A
B
A
00005
A
A
B
A
0012
A
A
A
, http://www.100md.com
B
00014
A
A
B
D
00010
A
C
C
E
00195
B
A
, 百拇医药
D
D
00480
A
D
C
F
00282
A
A
A
A
00481
A
, 百拇医药
A
A
A
00457
A
A
A
A
注:A~F分别为不同的DNA带型,记录方法如下:每一块凝胶按自左至右的顺序依次记录各菌株的带型,第1泳道带型记为A;第2泳道带型与第1泳道相同者记为A,不相同则记为B;第3泳道带型和第1泳道相同者记为A,和第2泳道相同者记为B,否则记为C,依次类推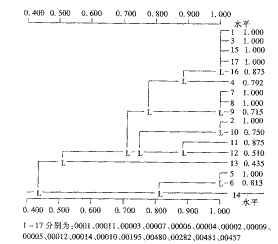
, 百拇医药
1~17分别为:0001、00011、00003、00007、00006、00004、00002、00009、00005、00012、00014、00010、00195、00480、00282、00481、00457
图2甄氏外瓶霉聚类分析树状图
讨论
甄氏外瓶霉是一组较为复杂的群体,目前认为其包括3个变种:E.jeanselme ivar.jeanselmei;E.jeanselme ivar.heteromorpha;E.jeanselme ivar.lecanii-corni。外抗原实验显示有3种血清型;DNA杂交将其分为6型[5],其中1、2、3型为血清Ⅰ型,4型为血清Ⅱ型,5、6型与血清Ⅲ型相一致;线粒体DNARFLP分析则将45株甄氏外瓶霉分为15型,但作者未标明与血清型的关系[6]。从以上实验可见,甄氏外瓶霉并非单一的种,而是种的复合体,分型较为复杂。
, http://www.100md.com
核糖体DNAPCR分型早已用于真菌种及种以上水平的分类,其转录间隔区(ITS)在核糖体DNA中进化较快,可以在同一属间甚至群体间发生变化。资料表明,ITSⅡ区在种内变异性较低(<3%),种间变异性较高(约22%)[7,8]。引物ITS3位于5.8SrDNA5′端,ITS4位于25SrDNA5′端,用该对引物得到的扩增片段包括5.8SrDNA大部分序列及完整的ITSⅡ区,约为330bp左右。
本实验中,15株菌可分为6型,多数菌株(13株)具有较近的亲缘关系(SM>0.7),型内相似程度较高,各变种间差异较大,SM值在0.5左右,与种间SM值相似。此结果还与18SrDNA分型和ITS1-ITS4分析的结果一致[9,10]。本研究支持3个变种应分属于不同种的观点。我们还发现同属变种E.jeanselme ivar.heteromorpha的BMU00457与BMU00010酶切带型差异较大(SM=0.51),前者与模式株BMU00001具有较高的亲缘关系。对于该变种的划分,还有商榷的余地;BMU00195分离自我国1例神经系统暗色丝孢霉病患者的病损组织,加拿大和美国学者将其鉴定为E.jeanselme ivar.lecanii-corni。1997年,ATCC将其收录为E.jeanselme ivar.lecanii-corni,并编入其菌种目录(编号为ATCC201307)。本实验中,此菌与同一变种的BMU00480(ATCC12734)差异极大,4种酶切图谱有3种不同,与其它菌株相似程度亦较低。在包括其它菌种的所有受试菌株中,只有该ITSⅡ序列能被HinFⅠ识别,提示该菌有可能为新变种或者为新种,但尚有待于进一步深入研究其多方面的特性。
, http://www.100md.com
另外,从实验结果还可以看出,种群分布似乎与菌种来源存在某种关系,Ⅱ、Ⅵ型同为环境分离株;Ⅲ、Ⅳ型为临床分离株,其中BMU00014表现出与BMU00010(E.jeanselme ivar.heteromorpha)较高的相似性(SM=0.875),可能为同一变种,但有待于进一步证实。BMU00009、BMU00005酶切带型相同,而前者过去命名为高氏瓶霉(Phialophoragougerotii),后者命名为甄氏瓶霉(Phialophorajeanselmei),对于此2种菌,以往经过分子方面的研究已证实为同一甄氏变种,本研究再次证实了上述结论。
志谢日本千叶大学真菌医学研究中心为本实验提供了部分受试菌株;中国科学院微生物研究所黄河研究员、刘小勇硕士在实验方法上给予了帮助
参考文献
[1]Elizabeth JS, Arthur JC, Farrar WE. Phaeohyphomycosis due to Exophiala species: clinical spectrum of diseases in humans. Clin Infect Dis, 1992,15:639- 644.
, 百拇医药
[2]Li RY, Wang DL. Studies on phaeohyphomycosis and its causative agents in China. Jpn J Med Mycol, 1996,37:135- 141.
[3]朱衡,瞿峰,朱立煌.利用氯化苄提取适用于分子生物学分析的真菌DNA.真菌学报,1994,13:34-40.
[4]Innis MA, Gelfland DH, Sninsky JJ, et al. PCR protocols. San Diego: Academic Press, 1990.315- 322.
[5]Matsuda M, Naka W, Tajima S, et al. Deoxyribonucleic acid hybridization studies of Exophiala dermatitidis and E. jeanselmei. Microbiol Immunol, 1989,33:631- 639.
, 百拇医药
[6]Kawasaki M. Mitochondrial DNA analysis of Exophiala jeanselmei and E.dermatitides. Mycopathologia,1990,110:107- 112.
[7]Hibbett DS, Vilgalys R. Evolutionships of Leninus to the polyporaceae: evidence from restriction analysis of enzymatically amplified ribosomal DNA. Mycologia, 1991,83:425- 439.
[8]Hibbett DS, Vilgalys R. Polygenetic relationships of the basidiomyces genus Lentium inferred from molecular and morphological character. Syst Biol, 1993,18:409- 433.
, http://www.100md.com
[9]Uijthof JMJ, de Hoog GS. PCR- ribotyping of type isolates of currently accepted Exophiala and Phaeococcomyces species. Antonie Van Leeuwenhoek, 1995,68:35- 42.
[10]Uijthof JMJ. Relationships with the black genus Exophiala based on ITS1 sequences. Mycol Res, 1996,100:1265- 1271.
(收稿日期:1999-10-18), 百拇医药
单位:100034 北京大学第一医院皮肤科
关键词:甄氏外瓶霉;随机扩增多态DNA技术
中华皮肤科杂志000503
【摘要】目的对不同来源的甄氏外瓶霉进行分析,为建立更为合理的分型方法提供依据。方法以ITS3和ITS4为引物,对17株甄氏外瓶霉的核糖体DNA转录间隔区(ITSⅡ)进行扩增,4种内切酶(HinFⅠ、MspⅠ、BsuRⅠ和RsaⅠ)酶切。结果ITSⅡ区扩增片段约为330bp,17个菌株可分为Ⅵ型,3株变种分属于不同的型,BMU00195带型独特,有可能为新变种或新种;种群关系与来源显示出一定的相关性。结论PCR-RFLP用于甄氏外瓶霉的分型研究较为可靠,对于确定有争议菌种的分类学地位具有重要的意义。
, http://www.100md.com
PCR-ribotyping of Exophiala jeanselmei
LI Dongming,LI Ruoyu,WANG Duanli,et al.
(Department of Dermatology, First Hospital, Peking University, Beijing 100034)
【Abstract】 Objective To analyze Exophiala jeanselmei strains recovered from different sources and to establish a better method for typing the strains in this species. Methods The ribosomal internal transcribed spacer regions (ITSⅡ ) of 17 isolates Exophiala jeanselmei were amplified with primer ITS3 and ITS4 and digested with four restriction enzymes (HinFⅠ , MspⅠ , BsuRⅠ and RsaⅠ ). Results The amplicon length of the ITSⅡ was about 330bp, and 6 types of characteristic patterns for the species were found after restriction analysis. The variants of E. jeanselmei were unrelated and should therefore be maintained as separate species. The unique patterns of isolate BMU00195 were shown,which seems probably to be a new variant or even a new species. Conclusions PCR- RFLP is useful for the typing and identification of Exophiala jeanselmei, which is important in confirming the taxonomic position of a complex species.
, 百拇医药
【Key words】 Exophiala jeanselmei Random amplified polymorphic DNA technique
外瓶霉(Exophialaspp,E)为暗色丝孢霉病的主要致病菌之一,而甄氏外瓶霉(E.jeanselmei)是外瓶霉属中致病性较强的一个种,易侵犯皮肤、上消化道及心血管系统,且病灶可血行播散[1,2]。该种内各亚种间变异性较大,且侵袭力、毒力均有差别,对药物的反应性亦不同。由于该类真菌形态及生理生化性质差异较小,传统的分型方法往往不能满足需要,分子生物学技术的发展为从基因水平上揭示真菌的亲缘关系提供了新的手段。近年来,核糖体DNA(rDNA)内转录间隔区(internaltranscribedspacer,ITS)已被应用于外瓶霉及其相近属种的分类中。本实验在国际上首次将PCR-RFLP应用于甄氏外瓶霉的种内分型研究,以期为建立更为合理的分类体系提供依据。
材料和方法
, 百拇医药
一、标本来源
包括甄氏外瓶霉及其变种共17株,均为北京大学真菌和真菌病研究中心保存菌株。
二、实验方法
————————
(一)DNA提取:将实验菌株接种于PDA,27℃培养2周,收集菌丝体约0.1g,用氯化苄法提取DNA[3],并在质量分数为0.8%琼脂糖凝胶上检测DNA的量和浓度。
(二)PCR扩增:应用引物ITS3和ITS4[4](华美公司合成)对ITSⅡ进行扩增。50μL反应体系含10mmolTris·HCl,1.5mmolMgCl,800μmol4dNTP,0.4μmolITS3,0.4μmolITS4TaqDNA聚合酶1U。反应条件:94℃变性1min,52℃退火50s,72℃延伸1min20s,35个循环。
, 百拇医药
(三)酶切:将PCR产物分别加入MspⅠ、BsuRⅠ、RsaⅠ和HinFⅠ37℃孵育3h,纯化后用质量分数为2%琼脂糖凝胶分离,以puc19DNA经MspⅠ彻底水解后的产物(MBI公司生产)作为分子量标记。紫外灯下照相,并在UV凝胶成象分析系统中成象。
(四)统计分析:应用单匹配系数(Singlematchingcoefficient,SM)表示物种的亲缘关系,计算方法为:SM=a/(a+b),式中a为两菌共有的“1”和“0”的条带数,“b”为非共有的条带数。菌株间的相似性为:SM×%。将每个菌的“1”和“0”输入,经Ntsys软件分析得到单匹配系数矩阵图,用该软件的UPGMA聚类分析法生成表示菌株间亲缘关系的树状图。

图1甄氏外瓶霉ITSⅡ区酶切带型图
结果
, http://www.100md.com
各菌株ITS间区扩增片段约为330bp,4种内切酶酶切后带型见表1。HinFI只对北京大学真菌和真菌病研究中心(BMU)00195(E.jeanselme ivar.lecanii-corni)有一个识别位点,MapⅠ及RsaⅠ对多数菌株均具有一个以上的切点,BsuRⅠ则只对半数以上菌株的ITS区有识别位点(图1)。
多种内切酶酶切带型综合聚类分析见图2,树状图主要分为6支,第1支包括模式株在内共6株菌,其中模式株BMU00001与标准株BMU00282、BMU00481、BMU00457及BMU00011、BMU00003带型完全相同(SM=1),与BMU00007相似程度亦较高(SM=0.875);第2支中3株菌为自然分离株(SM=1);第3支中分离自患者的BMU00007及BMU00012显示出高度相似性(SM=1);在第4支中,临床分离株BMU00014与BMU00010(E.jeanselme ivar.heteromorpha)相似程度较高(SM=0.875);分离自患者的BMU00195(E.jeanselme ivar.lecanii-corni)带型独特,单独成为第5支。第6支包括2株环境分离株和1株E.jeanselme ivar.lecanii-corni(BMU00480)。3株变种间相似程度较低(SM<0.51),而E.jeanselme ivar.heteromorph(BMU00457)却与标准菌株之间存在高度相似性。
, http://www.100md.com
表1甄氏外瓶霉ITSⅡ间区酶切带型表
菌号(BMU)
HinFⅠ
MspⅠ
BsuRⅠ
RsaⅠ
00001
A
A
A
A
00011
A
, 百拇医药
A
A
B
00003
A
A
A
A
00007
A
A
A
C
00006
, 百拇医药
A
B
B
A
00004
A
B
B
A
00002
A
A
B
A
, 百拇医药
00009
A
A
B
A
00005
A
A
B
A
0012
A
A
A
, http://www.100md.com
B
00014
A
A
B
D
00010
A
C
C
E
00195
B
A
, 百拇医药
D
D
00480
A
D
C
F
00282
A
A
A
A
00481
A
, 百拇医药
A
A
A
00457
A
A
A
A
注:A~F分别为不同的DNA带型,记录方法如下:每一块凝胶按自左至右的顺序依次记录各菌株的带型,第1泳道带型记为A;第2泳道带型与第1泳道相同者记为A,不相同则记为B;第3泳道带型和第1泳道相同者记为A,和第2泳道相同者记为B,否则记为C,依次类推

, 百拇医药
1~17分别为:0001、00011、00003、00007、00006、00004、00002、00009、00005、00012、00014、00010、00195、00480、00282、00481、00457
图2甄氏外瓶霉聚类分析树状图
讨论
甄氏外瓶霉是一组较为复杂的群体,目前认为其包括3个变种:E.jeanselme ivar.jeanselmei;E.jeanselme ivar.heteromorpha;E.jeanselme ivar.lecanii-corni。外抗原实验显示有3种血清型;DNA杂交将其分为6型[5],其中1、2、3型为血清Ⅰ型,4型为血清Ⅱ型,5、6型与血清Ⅲ型相一致;线粒体DNARFLP分析则将45株甄氏外瓶霉分为15型,但作者未标明与血清型的关系[6]。从以上实验可见,甄氏外瓶霉并非单一的种,而是种的复合体,分型较为复杂。
, http://www.100md.com
核糖体DNAPCR分型早已用于真菌种及种以上水平的分类,其转录间隔区(ITS)在核糖体DNA中进化较快,可以在同一属间甚至群体间发生变化。资料表明,ITSⅡ区在种内变异性较低(<3%),种间变异性较高(约22%)[7,8]。引物ITS3位于5.8SrDNA5′端,ITS4位于25SrDNA5′端,用该对引物得到的扩增片段包括5.8SrDNA大部分序列及完整的ITSⅡ区,约为330bp左右。
本实验中,15株菌可分为6型,多数菌株(13株)具有较近的亲缘关系(SM>0.7),型内相似程度较高,各变种间差异较大,SM值在0.5左右,与种间SM值相似。此结果还与18SrDNA分型和ITS1-ITS4分析的结果一致[9,10]。本研究支持3个变种应分属于不同种的观点。我们还发现同属变种E.jeanselme ivar.heteromorpha的BMU00457与BMU00010酶切带型差异较大(SM=0.51),前者与模式株BMU00001具有较高的亲缘关系。对于该变种的划分,还有商榷的余地;BMU00195分离自我国1例神经系统暗色丝孢霉病患者的病损组织,加拿大和美国学者将其鉴定为E.jeanselme ivar.lecanii-corni。1997年,ATCC将其收录为E.jeanselme ivar.lecanii-corni,并编入其菌种目录(编号为ATCC201307)。本实验中,此菌与同一变种的BMU00480(ATCC12734)差异极大,4种酶切图谱有3种不同,与其它菌株相似程度亦较低。在包括其它菌种的所有受试菌株中,只有该ITSⅡ序列能被HinFⅠ识别,提示该菌有可能为新变种或者为新种,但尚有待于进一步深入研究其多方面的特性。
, http://www.100md.com
另外,从实验结果还可以看出,种群分布似乎与菌种来源存在某种关系,Ⅱ、Ⅵ型同为环境分离株;Ⅲ、Ⅳ型为临床分离株,其中BMU00014表现出与BMU00010(E.jeanselme ivar.heteromorpha)较高的相似性(SM=0.875),可能为同一变种,但有待于进一步证实。BMU00009、BMU00005酶切带型相同,而前者过去命名为高氏瓶霉(Phialophoragougerotii),后者命名为甄氏瓶霉(Phialophorajeanselmei),对于此2种菌,以往经过分子方面的研究已证实为同一甄氏变种,本研究再次证实了上述结论。
志谢日本千叶大学真菌医学研究中心为本实验提供了部分受试菌株;中国科学院微生物研究所黄河研究员、刘小勇硕士在实验方法上给予了帮助
参考文献
[1]Elizabeth JS, Arthur JC, Farrar WE. Phaeohyphomycosis due to Exophiala species: clinical spectrum of diseases in humans. Clin Infect Dis, 1992,15:639- 644.
, 百拇医药
[2]Li RY, Wang DL. Studies on phaeohyphomycosis and its causative agents in China. Jpn J Med Mycol, 1996,37:135- 141.
[3]朱衡,瞿峰,朱立煌.利用氯化苄提取适用于分子生物学分析的真菌DNA.真菌学报,1994,13:34-40.
[4]Innis MA, Gelfland DH, Sninsky JJ, et al. PCR protocols. San Diego: Academic Press, 1990.315- 322.
[5]Matsuda M, Naka W, Tajima S, et al. Deoxyribonucleic acid hybridization studies of Exophiala dermatitidis and E. jeanselmei. Microbiol Immunol, 1989,33:631- 639.
, 百拇医药
[6]Kawasaki M. Mitochondrial DNA analysis of Exophiala jeanselmei and E.dermatitides. Mycopathologia,1990,110:107- 112.
[7]Hibbett DS, Vilgalys R. Evolutionships of Leninus to the polyporaceae: evidence from restriction analysis of enzymatically amplified ribosomal DNA. Mycologia, 1991,83:425- 439.
[8]Hibbett DS, Vilgalys R. Polygenetic relationships of the basidiomyces genus Lentium inferred from molecular and morphological character. Syst Biol, 1993,18:409- 433.
, http://www.100md.com
[9]Uijthof JMJ, de Hoog GS. PCR- ribotyping of type isolates of currently accepted Exophiala and Phaeococcomyces species. Antonie Van Leeuwenhoek, 1995,68:35- 42.
[10]Uijthof JMJ. Relationships with the black genus Exophiala based on ITS1 sequences. Mycol Res, 1996,100:1265- 1271.
(收稿日期:1999-10-18), 百拇医药