鼠疫耶尔森氏菌16S-23SrRNA基因间区分析
作者:魏建春 俞东征 刘梅 周艳 蔡虹
单位:中国预防医学科学院 流行病学微生物学研究所,北京 102206
关键词:鼠疫耶尔森氏菌;PCR;rRNA基因
中国地方病学杂志990405 摘要 目的 发展一种快速准确的鉴定鼠疫菌的方法。方法 PCR反应加限制性内切酶消化的方法。结果 对来自不同疫源地的103株鼠疫菌16S-23S rRNA基因间区进行扩增,均可扩出两条长为1 146bp及1 090bp的片段,扩增产物用限制性内切酶Taq I,Msp I消化后的酶切图谱相同。而对照菌株小肠结肠炎耶尔森氏菌,鼠伤寒沙门氏菌,大肠杆菌,痢疾杆菌,霍乱弧菌的扩增产物及酶切图谱与鼠疫菌均不相同。结论 鼠疫菌16S-23S rRNA基因间区高度同源,基本为两种类型,长度分别为1 146bp及1 090bp,其他菌株与其明显不同;该方法将有助于快速准确的鉴定鼠疫菌。
, 百拇医药
中图分类号 R516.8 [文献标识码]A [文章编号]1000-4955(1999)04-0257-04
Study on 16S-23S rRNA spacer region of Y.pestis
WEI Jian-chun,YU Dong-zheng,LIU Mei,et al
(Institute of Epidemiology and Microbiology,CAPM,Beijing 102206,China)
abstract Objective To develop a method for the indentification of Y.pestis.Methods PCR amplification and endonucleases digestion.Results The PCR amplification of 16S-23S rRNA spacer region was done on 103 Y.pestis strains that isolated from 9 different natural foci.Two fragments of 1 146bp and 1 090bp were obtained in all strains.The amplicon from each strain was digested with two endonucleases (Taq I,Msp I),and all isolates were displayed an identical pattern.Some control strains of E.coli,Y.enterocolitica,Salmonella typhimurium,Vibrio cholera,Shigella flexneri were analyzed by the approach,it was found that the amplified products and restriction patterns were different from that of Y.pestis.Conclusions The 16S-23S rRNA spacer regions are identical in all Y.pestis strains,and different from the other control strains;the approach will be helpful to the identification of Y.pestis.
, 百拇医药
Key words Y.pestis;PCR;rRNA gene
鼠疫菌的一般检查方法包括显微镜检查,培养,鼠疫噬菌体裂解试验及动物试验等4个步骤,通称4步检查或4步诊断。由于鼠疫菌的传播快,毒力强高居各类传染病之首,因此要求对鼠疫的最终判定不得超过96~108小时,对主要材料应在24小时内根据快速诊断提出预报。一般根据4步检查即可判定鼠疫,但因有的鼠疫菌形态不典型,噬菌体裂解试验也存在假阳性及假阴性现象,故可能出现误判或漏判[1]。而动物试验则需要较长的时间,因此我们希望发展一种快速准确的鉴定鼠疫菌的方法。为此,我们在实验室中对鼠疫菌16S-23S rRNA基因间段进行了分析,现将结果报告如下。
1 材料与方法
1.1 试验菌株
1.1.1 菌株来源:试验所用103株鼠疫菌由青海省鼠疫菌种保存中心提供,菌株来源见表1。EV菌为本试验室保存菌株,4株霍乱弧菌由本所02室提供,6株鼠伤寒沙门氏菌,8株小肠结肠炎耶尔森氏菌由本所免疫室提供,2株痢疾杆菌,6株大肠杆菌由本所微生物室提供。
, http://www.100md.com
1.1.2 菌株培养:所有菌株均接种于普通LB培养基中,鼠疫菌于28℃,其它菌于37℃过夜培养。
表1 103株鼠疫菌菌株来源 疫源地名称
分离地区
菌株数
松辽平源达乌尔黄鼠疫源地
内蒙古高原长爪沙鼠疫源地
锡林格勒高原布氏田鼠疫源地
天山山地灰旱獭疫源地
帕米尔高原长尾旱獭疫源地
甘宁黄土高原阿拉善黄鼠疫源地
, 百拇医药
青藏高原喜马拉雅旱獭疫源地
滇西北山地大绒鼠疫源地
滇西居民区黄胸鼠疫源地
白城 长岭 扶余 翁牛特 敖汗 明太
康保 西苏 鄂托克
苏尼特左旗 阿巴哈那尔 阿巴嘎
玛纳斯 呼图壁 昌吉 乌苏 精河 尼勒克 伊宁
阿图什 乌恰
海源 西吉 固源 会宁
那曲 共和 都兰 仲巴 门源 祁连肃南 和田 若羌 洛浦
, 百拇医药
剑川
弥渡 蒲田 祥云 陇川 瑞丽 保山
10
5
5
15
5
10
33
5
15
1.2 主要设备及试剂
1.2.1 主要设备:高速台式离心机(TGL-16C型),PCR扩增仪(中科院遗传所,TC-96AE型),水平及垂直电泳装置(北京东方仪器厂),紫外检测仪(永嘉上塘教学仪器厂)。
, 百拇医药
1.2.2 主要试剂:TaqDNA多聚酶购自中科院遗传所,蛋白酶K,dNTP,限制性内切酶Taq I,Msp I,分子量标准λDNA/HindⅢ+EcoR I,pGEM-7zf(+)/HaeⅢ等购自华美生物工程公司,其它均为常规试剂。
1.3 染色体DNA提取方法:参照参考文献[2]。
1.4 PCR扩增
1.4.1 引物设计:根据大肠杆菌16S rRNA,小肠结肠炎耶尔森氏菌23S rRNA基因中高度保守区设计了一对16S rRNA基因,23S rRNA基因的反向引物,其序列为:
16S rRNA基因反向引物A2:5′TGC GGC TGG ATC ACC TCC TT3′。
23S rRNA基因反向引物C1:5′CCC ATT ATA CAA AAG GTA CGC A3′。
, 百拇医药
引物由中国科学院微生物所合成。
1.4.2 PCR扩增:总反应体积为50μl,在0.5ml Eppendorf管中依次加入下列成分;三蒸水34.5μl,10×buffer5μl(1×buffer),dNTP混合物4μl(0.2mM/Each),引物A2,C1各2μl(1μM),模板2μl,TaqDNA聚合酶0.5μl(1.5U)。混匀离心,加等体积液体石蜡封闭。94℃变性8分钟,然后进行下列30个循环,94℃1 分钟,58℃1分钟,72℃2分钟,最后72℃延伸5分钟。
1.4.3 PCR扩增产物的检测:取7μl反应产物,加2μl电泳载样液,1%琼脂糖凝胶,6V/cm电泳2小时,经溴化乙锭染色后在紫外检测仪上观察结果。
1.4.4 PCR扩增产物的酶切:取9μl扩增产物,加1μl 10×buffer,4~7U限制性内切酶,适当温度条件下消化3小时。
, 百拇医药
1.4.5 聚丙烯酰胺凝胶电泳检测:在10μl酶切产物中加2μl电泳载样缓冲液,取3~4μl于8%聚丙烯酰胺凝胶上电泳,电压6V/cm,溴酚蓝移至胶底时终止电泳。银染后拍照。
2 结果
2.1 16S rRNA,23S rRNA基因间区的PCR扩增:被试的103株鼠疫菌用A2,C1两引物均可扩出两条长约1 146bp,1 090bp的片段,见图1。对大肠杆菌,痢疾杆菌,小肠结肠炎耶尔森氏菌,霍乱弧菌,鼠伤寒沙门氏菌等几种细菌染色体的16S rRNA-23S rRNA基因间区进行扩增,结果见图2。几种细菌该基因区段均可被扩增出来,大小在1kb左右,在1%琼脂糖凝胶上电泳与鼠疫菌均不相同。
图1 鼠疫菌PCR扩增产物1%琼脂糖凝胶电泳图
, 百拇医药
两边为分子量标准λDNA/EcoR I+HindⅢ,1~13依次为:EV 44001 490009 84038 74006 46501 38003 45056 41002 12004 34002 49002 83007
14为空白对照.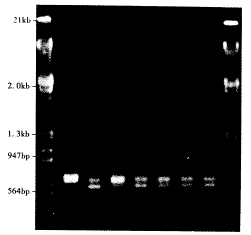
图2 对照菌株PCR扩增产物1%琼脂糖凝胶电泳图
两边为分子量标准λDNA/EcoR I+HindⅢ,1为EV菌,2~3为痢疾杆菌,4~7为大肠杆菌.
2.2 扩增产物的限制性内切酶酶切分析:本实验应用2种限制性内切酶Taq I,Msp I对扩增产物进行酶切,结果发现不同鼠疫菌株间的酶切图谱一致,见图2。选用Taq I对其他几种细菌进行酶切分析,结果见图4,图5。由图中可以看出,实验所用其他菌株的酶切图谱与鼠疫菌均明显不同。
, 百拇医药
图3 鼠疫菌扩增产物Taq I酶切的8%聚丙烯酰胺凝胶电泳图
两边为分子量标准pGEM-7zf(+)/HaeⅢ,1~12依次为:13011 30007 32002 34003 41002 74010 103001 124001 126001 74010 46501 12007.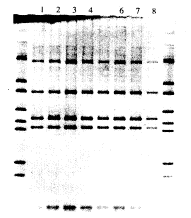
图4 鼠疫菌扩增产物Msp I酶切的8%聚丙烯酰胺凝胶电泳图
两边为分子量标准pGEM-7zf(+)/HaeⅢ,1~8依次为:01074 12006 32002 46501 74002 83006 91003 125002.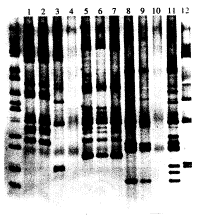
图5 对照菌株扩增产物Taq I酶切的8%聚丙烯酰胺凝胶电泳图
, http://www.100md.com
加样顺序为:分子量标准pGEM-7zf(+)/HaeⅢ 1~2为鼠伤寒沙门氏菌,3~7为小肠结肠炎耶尔森氏菌,8~11为霍乱弧菌,12为EV菌.
3 讨论
随着分子生物学技术的发展,在基因水平上对细菌进行鉴定,分型的方法越来越多,其中PCR方法由于其快速简便而得到了越来越多的应用,已有人把该方法用于鼠疫菌的检测和鉴定。Cambell-J于1993年使用一种快速特异的巢式PCR方法来鉴定鼠疫菌[3],Tsukano-H等人则发展了一种多引物PCR方法检测和鉴定鼠疫菌[4],我们采用了一种PCR加酶切的方法(即PCR-based RFLP)对分离自不同疫源地分属17个生态型的103株鼠疫菌的16S-23 rRNA基因间区进行了分析。
16S-23S rRNA基因间区的扩增需要有合适的引物,引物序列应位于16S和23S rRNA基因中的高度保守区,目前已知有许多细菌种属的16S rRNA和23S rRNA基因的全长序列已有报道,这就为引物的设计提供了方便。本研究即根据16S及23S rRNA基因中的一段高度保守序列设计了引物,结果表明该引物对鼠疫菌是非特异性的,实验所用鼠疫菌及其他菌株均可用该引物扩出长约1kb的片段。
, 百拇医药
103株鼠疫菌的16S-23S rRNA基因间区扩增产物在琼脂糖凝胶电泳上几乎完全相同,难以区分开来,为进一步分析该基因区段在不同鼠疫菌间同源性的程度,我们选用两种限制性内切酶Taq I,Msp I对该区段进行了分析,电泳结果发现103株鼠疫菌的酶切图谱一致。这说明我们所用的两种酶在16S-23S rRNA基因间区中的酶切位点是高度保守的。
由于鼠疫菌16S-23S rRNA基因间区酶切图谱的一致性使我们想了解鼠疫菌的这一特征是否具有种属特异性,为此,我们又对霍乱弧菌,小肠结肠炎耶尔森氏菌,鼠伤寒沙门氏菌,痢疾杆菌,大肠杆菌的16S-23S rRNA基因间区进行了分析。所有对照菌株均可扩出长约1kb的片段,但由于rRNA操纵子在不同种属细菌中拷贝数,长度均可能不同,因此不同细菌的扩增产物在琼脂糖凝胶电泳上也可看出差别,片段大小稍有不同,且扩出的片段数目也有不同,有的只有一条带,有的则可见两三条带,不同细菌的扩增产物与鼠疫菌均有差别。为进一步验证不同种属细菌间该基因区段的种属特异性,我们也对这些细菌的扩增产物进行了酶切分析。结果表明所有对照细菌的酶切图谱与鼠疫菌的均明显不同。说明鼠疫菌的这一特征具有种属特异性,可用于对鼠疫菌的鉴定工作。
, 百拇医药
作者简介:魏建春,女,1972年生,硕士研究生
参考文献
[1] 纪树立.鼠疫[M].北京:人民卫生出版社,1988.
[2] Robert J,Owen and Pauline Borman.A rapid biochemical method for purifying high molecular weight bacterial chromosomal DNA for restriction enzyme analysis [J].Nucleic Acid Reserch,1987,15(8):3631.
[3] Cambell J,Lowe J,Walz S and Ezzell JW.Rapid and specific identification of Yersinia pestis by using a nested polymerase chain reaction procedure [J].Clin J.Microbiol,1993,31(3):1758-1759.
[4] Tsukano H,Itoh K,Suzuki S,et al.Detection and identification of Yersinia pestis by using multiplex primers[J].Microbiol Immunol,1996,40(10):773-775.
收稿日期1999-03-10;修订日期1999-03-26, 百拇医药
单位:中国预防医学科学院 流行病学微生物学研究所,北京 102206
关键词:鼠疫耶尔森氏菌;PCR;rRNA基因
中国地方病学杂志990405 摘要 目的 发展一种快速准确的鉴定鼠疫菌的方法。方法 PCR反应加限制性内切酶消化的方法。结果 对来自不同疫源地的103株鼠疫菌16S-23S rRNA基因间区进行扩增,均可扩出两条长为1 146bp及1 090bp的片段,扩增产物用限制性内切酶Taq I,Msp I消化后的酶切图谱相同。而对照菌株小肠结肠炎耶尔森氏菌,鼠伤寒沙门氏菌,大肠杆菌,痢疾杆菌,霍乱弧菌的扩增产物及酶切图谱与鼠疫菌均不相同。结论 鼠疫菌16S-23S rRNA基因间区高度同源,基本为两种类型,长度分别为1 146bp及1 090bp,其他菌株与其明显不同;该方法将有助于快速准确的鉴定鼠疫菌。
, 百拇医药
中图分类号 R516.8 [文献标识码]A [文章编号]1000-4955(1999)04-0257-04
Study on 16S-23S rRNA spacer region of Y.pestis
WEI Jian-chun,YU Dong-zheng,LIU Mei,et al
(Institute of Epidemiology and Microbiology,CAPM,Beijing 102206,China)
abstract Objective To develop a method for the indentification of Y.pestis.Methods PCR amplification and endonucleases digestion.Results The PCR amplification of 16S-23S rRNA spacer region was done on 103 Y.pestis strains that isolated from 9 different natural foci.Two fragments of 1 146bp and 1 090bp were obtained in all strains.The amplicon from each strain was digested with two endonucleases (Taq I,Msp I),and all isolates were displayed an identical pattern.Some control strains of E.coli,Y.enterocolitica,Salmonella typhimurium,Vibrio cholera,Shigella flexneri were analyzed by the approach,it was found that the amplified products and restriction patterns were different from that of Y.pestis.Conclusions The 16S-23S rRNA spacer regions are identical in all Y.pestis strains,and different from the other control strains;the approach will be helpful to the identification of Y.pestis.
, 百拇医药
Key words Y.pestis;PCR;rRNA gene
鼠疫菌的一般检查方法包括显微镜检查,培养,鼠疫噬菌体裂解试验及动物试验等4个步骤,通称4步检查或4步诊断。由于鼠疫菌的传播快,毒力强高居各类传染病之首,因此要求对鼠疫的最终判定不得超过96~108小时,对主要材料应在24小时内根据快速诊断提出预报。一般根据4步检查即可判定鼠疫,但因有的鼠疫菌形态不典型,噬菌体裂解试验也存在假阳性及假阴性现象,故可能出现误判或漏判[1]。而动物试验则需要较长的时间,因此我们希望发展一种快速准确的鉴定鼠疫菌的方法。为此,我们在实验室中对鼠疫菌16S-23S rRNA基因间段进行了分析,现将结果报告如下。
1 材料与方法
1.1 试验菌株
1.1.1 菌株来源:试验所用103株鼠疫菌由青海省鼠疫菌种保存中心提供,菌株来源见表1。EV菌为本试验室保存菌株,4株霍乱弧菌由本所02室提供,6株鼠伤寒沙门氏菌,8株小肠结肠炎耶尔森氏菌由本所免疫室提供,2株痢疾杆菌,6株大肠杆菌由本所微生物室提供。
, http://www.100md.com
1.1.2 菌株培养:所有菌株均接种于普通LB培养基中,鼠疫菌于28℃,其它菌于37℃过夜培养。
表1 103株鼠疫菌菌株来源 疫源地名称
分离地区
菌株数
松辽平源达乌尔黄鼠疫源地
内蒙古高原长爪沙鼠疫源地
锡林格勒高原布氏田鼠疫源地
天山山地灰旱獭疫源地
帕米尔高原长尾旱獭疫源地
甘宁黄土高原阿拉善黄鼠疫源地
, 百拇医药
青藏高原喜马拉雅旱獭疫源地
滇西北山地大绒鼠疫源地
滇西居民区黄胸鼠疫源地
白城 长岭 扶余 翁牛特 敖汗 明太
康保 西苏 鄂托克
苏尼特左旗 阿巴哈那尔 阿巴嘎
玛纳斯 呼图壁 昌吉 乌苏 精河 尼勒克 伊宁
阿图什 乌恰
海源 西吉 固源 会宁
那曲 共和 都兰 仲巴 门源 祁连肃南 和田 若羌 洛浦
, 百拇医药
剑川
弥渡 蒲田 祥云 陇川 瑞丽 保山
10
5
5
15
5
10
33
5
15
1.2 主要设备及试剂
1.2.1 主要设备:高速台式离心机(TGL-16C型),PCR扩增仪(中科院遗传所,TC-96AE型),水平及垂直电泳装置(北京东方仪器厂),紫外检测仪(永嘉上塘教学仪器厂)。
, 百拇医药
1.2.2 主要试剂:TaqDNA多聚酶购自中科院遗传所,蛋白酶K,dNTP,限制性内切酶Taq I,Msp I,分子量标准λDNA/HindⅢ+EcoR I,pGEM-7zf(+)/HaeⅢ等购自华美生物工程公司,其它均为常规试剂。
1.3 染色体DNA提取方法:参照参考文献[2]。
1.4 PCR扩增
1.4.1 引物设计:根据大肠杆菌16S rRNA,小肠结肠炎耶尔森氏菌23S rRNA基因中高度保守区设计了一对16S rRNA基因,23S rRNA基因的反向引物,其序列为:
16S rRNA基因反向引物A2:5′TGC GGC TGG ATC ACC TCC TT3′。
23S rRNA基因反向引物C1:5′CCC ATT ATA CAA AAG GTA CGC A3′。
, 百拇医药
引物由中国科学院微生物所合成。
1.4.2 PCR扩增:总反应体积为50μl,在0.5ml Eppendorf管中依次加入下列成分;三蒸水34.5μl,10×buffer5μl(1×buffer),dNTP混合物4μl(0.2mM/Each),引物A2,C1各2μl(1μM),模板2μl,TaqDNA聚合酶0.5μl(1.5U)。混匀离心,加等体积液体石蜡封闭。94℃变性8分钟,然后进行下列30个循环,94℃1 分钟,58℃1分钟,72℃2分钟,最后72℃延伸5分钟。
1.4.3 PCR扩增产物的检测:取7μl反应产物,加2μl电泳载样液,1%琼脂糖凝胶,6V/cm电泳2小时,经溴化乙锭染色后在紫外检测仪上观察结果。
1.4.4 PCR扩增产物的酶切:取9μl扩增产物,加1μl 10×buffer,4~7U限制性内切酶,适当温度条件下消化3小时。
, 百拇医药
1.4.5 聚丙烯酰胺凝胶电泳检测:在10μl酶切产物中加2μl电泳载样缓冲液,取3~4μl于8%聚丙烯酰胺凝胶上电泳,电压6V/cm,溴酚蓝移至胶底时终止电泳。银染后拍照。
2 结果
2.1 16S rRNA,23S rRNA基因间区的PCR扩增:被试的103株鼠疫菌用A2,C1两引物均可扩出两条长约1 146bp,1 090bp的片段,见图1。对大肠杆菌,痢疾杆菌,小肠结肠炎耶尔森氏菌,霍乱弧菌,鼠伤寒沙门氏菌等几种细菌染色体的16S rRNA-23S rRNA基因间区进行扩增,结果见图2。几种细菌该基因区段均可被扩增出来,大小在1kb左右,在1%琼脂糖凝胶上电泳与鼠疫菌均不相同。

图1 鼠疫菌PCR扩增产物1%琼脂糖凝胶电泳图
, 百拇医药
两边为分子量标准λDNA/EcoR I+HindⅢ,1~13依次为:EV 44001 490009 84038 74006 46501 38003 45056 41002 12004 34002 49002 83007
14为空白对照.

图2 对照菌株PCR扩增产物1%琼脂糖凝胶电泳图
两边为分子量标准λDNA/EcoR I+HindⅢ,1为EV菌,2~3为痢疾杆菌,4~7为大肠杆菌.
2.2 扩增产物的限制性内切酶酶切分析:本实验应用2种限制性内切酶Taq I,Msp I对扩增产物进行酶切,结果发现不同鼠疫菌株间的酶切图谱一致,见图2。选用Taq I对其他几种细菌进行酶切分析,结果见图4,图5。由图中可以看出,实验所用其他菌株的酶切图谱与鼠疫菌均明显不同。

, 百拇医药
图3 鼠疫菌扩增产物Taq I酶切的8%聚丙烯酰胺凝胶电泳图
两边为分子量标准pGEM-7zf(+)/HaeⅢ,1~12依次为:13011 30007 32002 34003 41002 74010 103001 124001 126001 74010 46501 12007.

图4 鼠疫菌扩增产物Msp I酶切的8%聚丙烯酰胺凝胶电泳图
两边为分子量标准pGEM-7zf(+)/HaeⅢ,1~8依次为:01074 12006 32002 46501 74002 83006 91003 125002.

图5 对照菌株扩增产物Taq I酶切的8%聚丙烯酰胺凝胶电泳图
, http://www.100md.com
加样顺序为:分子量标准pGEM-7zf(+)/HaeⅢ 1~2为鼠伤寒沙门氏菌,3~7为小肠结肠炎耶尔森氏菌,8~11为霍乱弧菌,12为EV菌.
3 讨论
随着分子生物学技术的发展,在基因水平上对细菌进行鉴定,分型的方法越来越多,其中PCR方法由于其快速简便而得到了越来越多的应用,已有人把该方法用于鼠疫菌的检测和鉴定。Cambell-J于1993年使用一种快速特异的巢式PCR方法来鉴定鼠疫菌[3],Tsukano-H等人则发展了一种多引物PCR方法检测和鉴定鼠疫菌[4],我们采用了一种PCR加酶切的方法(即PCR-based RFLP)对分离自不同疫源地分属17个生态型的103株鼠疫菌的16S-23 rRNA基因间区进行了分析。
16S-23S rRNA基因间区的扩增需要有合适的引物,引物序列应位于16S和23S rRNA基因中的高度保守区,目前已知有许多细菌种属的16S rRNA和23S rRNA基因的全长序列已有报道,这就为引物的设计提供了方便。本研究即根据16S及23S rRNA基因中的一段高度保守序列设计了引物,结果表明该引物对鼠疫菌是非特异性的,实验所用鼠疫菌及其他菌株均可用该引物扩出长约1kb的片段。
, 百拇医药
103株鼠疫菌的16S-23S rRNA基因间区扩增产物在琼脂糖凝胶电泳上几乎完全相同,难以区分开来,为进一步分析该基因区段在不同鼠疫菌间同源性的程度,我们选用两种限制性内切酶Taq I,Msp I对该区段进行了分析,电泳结果发现103株鼠疫菌的酶切图谱一致。这说明我们所用的两种酶在16S-23S rRNA基因间区中的酶切位点是高度保守的。
由于鼠疫菌16S-23S rRNA基因间区酶切图谱的一致性使我们想了解鼠疫菌的这一特征是否具有种属特异性,为此,我们又对霍乱弧菌,小肠结肠炎耶尔森氏菌,鼠伤寒沙门氏菌,痢疾杆菌,大肠杆菌的16S-23S rRNA基因间区进行了分析。所有对照菌株均可扩出长约1kb的片段,但由于rRNA操纵子在不同种属细菌中拷贝数,长度均可能不同,因此不同细菌的扩增产物在琼脂糖凝胶电泳上也可看出差别,片段大小稍有不同,且扩出的片段数目也有不同,有的只有一条带,有的则可见两三条带,不同细菌的扩增产物与鼠疫菌均有差别。为进一步验证不同种属细菌间该基因区段的种属特异性,我们也对这些细菌的扩增产物进行了酶切分析。结果表明所有对照细菌的酶切图谱与鼠疫菌的均明显不同。说明鼠疫菌的这一特征具有种属特异性,可用于对鼠疫菌的鉴定工作。
, 百拇医药
作者简介:魏建春,女,1972年生,硕士研究生
参考文献
[1] 纪树立.鼠疫[M].北京:人民卫生出版社,1988.
[2] Robert J,Owen and Pauline Borman.A rapid biochemical method for purifying high molecular weight bacterial chromosomal DNA for restriction enzyme analysis [J].Nucleic Acid Reserch,1987,15(8):3631.
[3] Cambell J,Lowe J,Walz S and Ezzell JW.Rapid and specific identification of Yersinia pestis by using a nested polymerase chain reaction procedure [J].Clin J.Microbiol,1993,31(3):1758-1759.
[4] Tsukano H,Itoh K,Suzuki S,et al.Detection and identification of Yersinia pestis by using multiplex primers[J].Microbiol Immunol,1996,40(10):773-775.
收稿日期1999-03-10;修订日期1999-03-26, 百拇医药