基于生物信息学的胰腺导管腺癌核心风险基因筛选和分析
模块,1材料与方法,1数据获取与处理,2差异表达分析,3GO和KEGG富集分析,4WGCNA的构建和分析,5PPI的构建和核心基因筛选,6核心风险基因的功能分析,7核心风险基因的临床诊断价值评价,2结果,1D
景晓莹,彭 亮,谢娜娜,吕环环,党 洁,2,马占兵,2(1.宁夏医科大学基础医学院,银川 750004;2.宁夏医科大学生育力保持教育部重点实验室,银川 750004)
胰腺导管腺癌(pancreatic ductal adenocarcinoma,PDAC)是一种恶性程度极高并具有高度侵袭性的消化道恶性肿瘤。因其5 年生存率不足7%,是恶性肿瘤中预后最差的[1-2]。PDAC 早期的症状隐匿、不典型[3],约80%的患者被确诊时为中晚期或出现转移,已错失最佳手术根治的窗口和机会。即使成功实施手术干预,术后12 个月内的复发率和转移率仍高达60%[4]。因此,准确有效的生物标记物筛选及其分子机制研究对于PDAC的诊断、治疗和不良预后改善具有十分重要的临床意义和研究价值。
加权基因共表达网络(weighted gene co-expression network analysis,WGCNA)已被广泛用于寻找各种癌症中的枢纽基因。癌症基因组图谱(the cancer genome atlas,TCGA)数据库中缺乏正常组织的PDAC 样本,差异表达数据不完整。因此,本研究整合基因型-组织表达数据库(genotypetissue expression,GTEx)数据库中正常对照组织的表达数据,有效地克服TCGA 数据库对照样本不足的问题。通过差异表达分析,获得PDAC 原发癌全面的转录组表达谱,并使用WGCNA、蛋白质相互作用网络(protein-protein interaction,PPI)网络分析结合表达生存分析,获得核心风险基因,以识别潜在准确的PDAC 生物标记物。
1 材料与方法
1.1 数据获取与处理
PDAC 组织的RNA-seq 数据来自TCGA,匹配的正常组织表达数据来自GTEx[5]。过滤去除基因表达值低于lcpm 剪切阈值80%以上的样本,通过filterByExpr 函数去除表达矩阵中不表达或低表达的基因。最后纳入共312 个样本,包含147 例原发肿瘤组织,165 例正常组织(图1)。
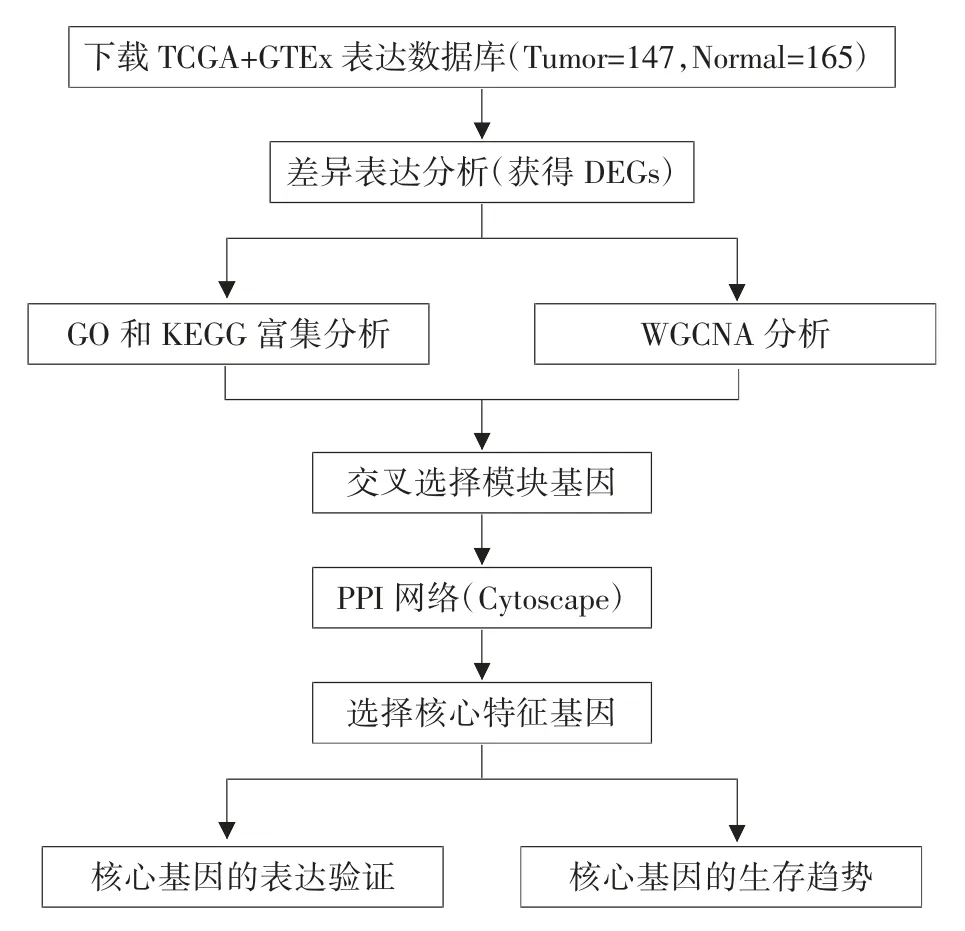
图1 数据下载、处理和分析流程
1.2 差异表达分析
使用edgeR 包筛选差异表达基因(differential expression genes ......
您现在查看是摘要页,全文长 10668 字符。